Aufgabenstellung
Wir untersuchen in dieser Aufgabe den Zusammenhang zwischen Attentional Load und Pupillengrösse. Wir verwenden dafür die Daten aus der Studie von Wahn et al. (2016). Es ist bekannt, dass die Grösse unserer Pupillen mit dem einfallenden Licht zusammenhängt. Es wird jedoch auch seit längerem vermutet, dass die Pupillengrösse mit kognitiven Prozessen zusammenhängt.
In dieser Studie untersuchten Wahn et al. (2016) den Einfluss des “attentional load” auf die Pupillengrösse von 20 Versuchspersonen anhand eines “Multiple Object Tracking” (MOT) Tasks.
Ein Beispieltrial ist in Grafik 1 dargestellt.
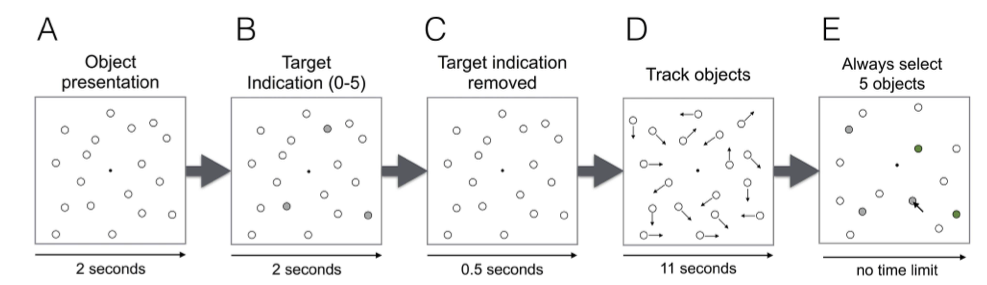
Figure 1: Beispieltrial eines Multiple Object Tracking Tasks. Versuchspersonen sahen zuerst 18 Objekte auf einem Bildschirm (A). Danach wurden zwischen 0 und 5 dieser Objekte anhand einer Markierung ausgewählt (B)—diese Objekte mussten visuell verfolgt werden. Die Markierungen verschwand wieder (C), und die Objekte fingen an, sich während 11 Sekunden zu bewegen (D). Am Ende des Trials mussten die Versuchspersonen angeben, welche Objekte sie verfolgt hatten. Die Anzahl der zu verfolgenden Objekte gilt als Mass für attentional load.
Üblicherweise wird in Pupillengrössen Exerimenten die Anteilsmässige Veränderung der Pupillengrössen in Prozent angegeben. Wir verwenden hier jedoch der Einfachheit halber die Rohwerte (gemittelt über beide Augen). Wir nehmen an, dass ein grösserer attentional load zu einer Erweiterung der Pupillen führt, und zwar nehmen wir hier an, dass der Zusammenhang zwischen attentional load und Pupillengrösse linear ist.
Daten laden
Wir laden zuerst die benötigten Packages, und die Daten. Die Personenvariable wird zu einem Faktor konvertiert. Ausserdem erstellen wir einen Faktor attentional_load, damit wir die Daten einfacher grafisch darstellen können.
pupilsize <- read_csv("https://raw.githubusercontent.com/kogpsy/neuroscicomplab/main/data/df_pupil_complete.csv") %>%
mutate(subj = as_factor(subj),
attentional_load = as_factor(load))
glimpse(pupilsize)
Rows: 2,228
Columns: 5
$ subj <fct> 701, 701, 701, 701, 701, 701, 701, 701, 701…
$ trial <dbl> 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, …
$ load <dbl> 2, 1, 5, 4, 0, 3, 0, 4, 2, 3, 5, 1, 4, 3, 0…
$ p_size <dbl> 1021.4086, 951.4349, 1063.9569, 913.4871, 6…
$ attentional_load <fct> 2, 1, 5, 4, 0, 3, 0, 4, 2, 3, 5, 1, 4, 3, 0…Die Variable p_size ist die Pupillengrösse, load ist der attentional load als numerische Variable mit den Werten 0-5. attentional_load ist der Faktor. subj ist die Versuchspersonennummer, und trial die Durchgangsnummer.
Wir haben pro Person eine unterschiedliche Anzahl Trials. Bei Vpn 701 handelt es sich wohl um Daten aus einer Pilotstudie.
pupilsize %>%
group_by(subj) %>%
count()
# A tibble: 20 x 2
# Groups: subj [20]
subj n
<fct> <int>
1 701 41
2 702 119
3 703 120
4 704 111
5 705 99
6 706 120
7 707 120
8 708 119
9 709 119
10 710 117
11 711 120
12 712 120
13 713 120
14 714 120
15 715 120
16 716 65
17 717 120
18 718 120
19 719 118
20 720 120Aufgaben
Aufgabe 1
Fassen Sie die Daten für jede Person pro attentional load Bedingung zusammen (Mittelwert, Standardfehler), und speichern Sie dies als Dataframe (benutzen Sie den
attentional_loadFaktor, d.h.group_by(subj, attentional_load)).Stellen Sie Mittelwert plus Fehlerbalken für jede Person in jeder Bedingung dar. Dies ist einfacher, als es sich anhört (benutzen Sie
facet_wrap(~subj, scales = "free_y")). Stellen Sieattentional_loadauf der X-Achse dar, die mittlere Pupillengrösse auf der Y-Achse.Beschreiben Sie in Worten (kurz) was sie hier sehen. Ist eine Tendenz ersichtlich? Ist diese bei jeder Person feststellbar? Gibt es Unterschiede zwischen den Personen?
by_subj %>%
ggplot(aes(attentional_load, mean)) +
geom_line(aes(group = 1), linetype = 3) +
geom_errorbar(aes(ymin = mean-se, ymax = mean+se),
width = 0.2, size=1, color="blue") +
geom_point(size = 2) +
facet_wrap(~subj, scales = "free_y")
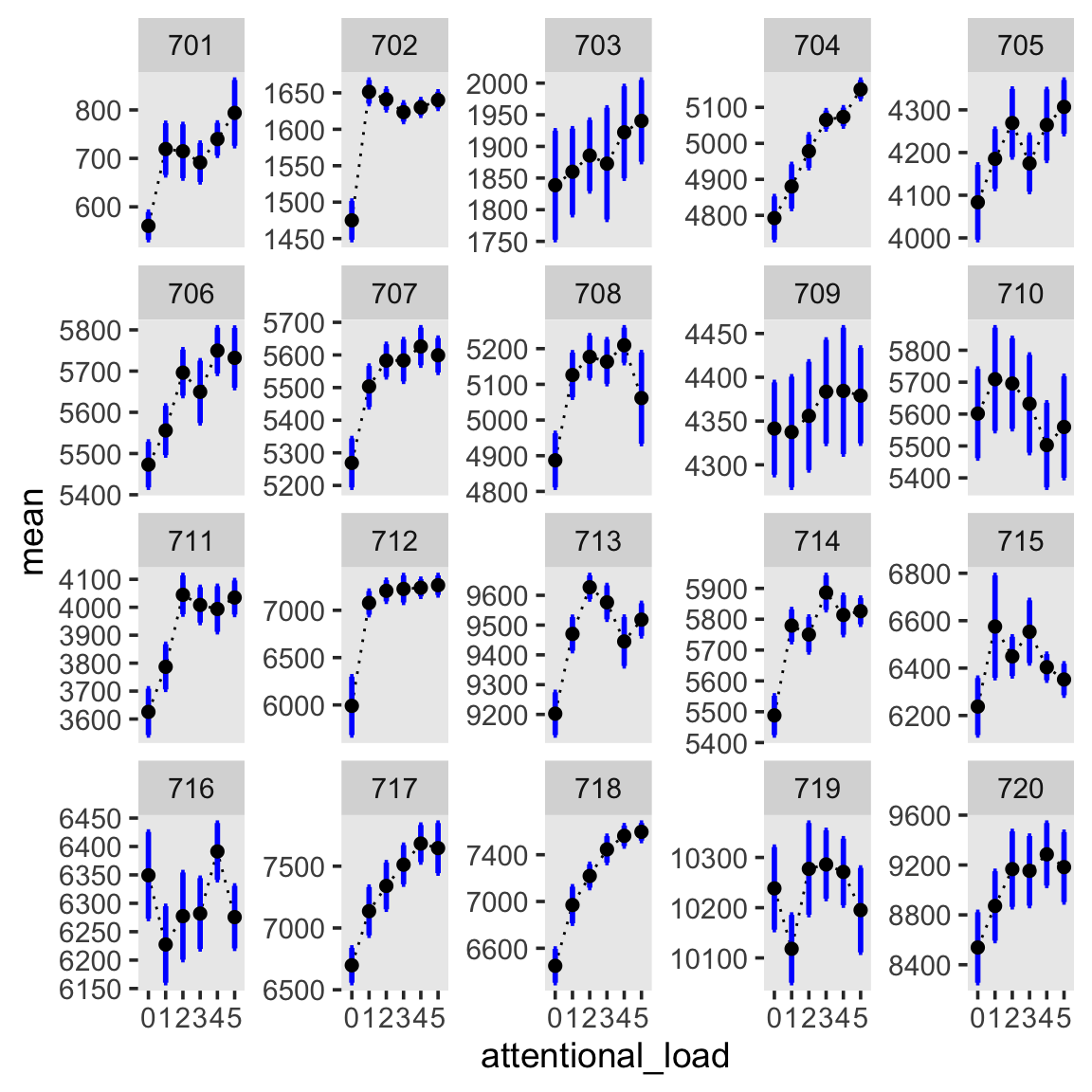
Es ist ersichtlich, dass die Pupillengrösse mit steigendem attentional load zunimmt, und zwar bei fast allen Personen. Ausnahmen sind Personen 710 und 716—bei diesen müsste man wohl genauer hinschauen. Zudem ist erkennbar, dass der Zusammenhang zwischen load und Pupillengrösse bei den meisten nicht linear zu sein scheint. Wir müssten dies auch untersuchen. In dieser Übung beschränken wir uns jedoch auf den linearen Zusammenhang.
Aufgabe 2
Sie sehen hier die mittlere Pupillengrösse pro attentional load Bedingung, aggregiert über Versuchspersonen. Die Fehlerbalken sind Standardfehler, welche die Messwiederholung berücksichtigen.
Beschreiben Sie in Worten (kurz) was sie hier sehen. Ist eine Tendenz ersichtlich?
agg <- Rmisc::summarySEwithin(by_subj,
measurevar = "mean",
withinvars = "attentional_load",
idvar = "subj",
na.rm = FALSE,
conf.interval = .95)
agg %>%
ggplot(aes(attentional_load, mean)) +
geom_line(aes(group = 1), linetype = 3) +
geom_errorbar(aes(ymin = mean-se, ymax = mean+se),
width = 0.2, size=1, color="blue") +
geom_point(size = 4)

Aggregiert über die Personen ist ein klarer Anstieg der Pupillengrösse in Abhängigkeit des attentional load erkennbar. Dieser Effekt scheint im Mittel nicht linear zu sein—der Anstieg ist am grössten zwischen 0 und 1, und flacht dann ab, so dass es scheinbar keinen Unterschied zwischen den attentional load 3-5 gibt.
Wir benutzen nun den Prädiktor load, um die Pupillengrösse mit einem Bayesianischen Multilevel vorherzusagen. Wir nehmen an, dass die Pupillengrösse bedingt normalverteilt ist (Diese Annahme ist vertretbar; obwohl eine Grösse niemals negativ sein darf, sind hier weit genug weg von 0).
Da die Personen sich ähnlich sind, sich jedoch in ihrer Pupillengrösse aufgrund ihrer Anatomie unterscheiden, benutzen wir ein partial-pooling Modell.
Annahmen:
- Die Outcome variable ist bedingt normalverteilt (oder die Fehler sind normalverteilt).
- Es gibt einen linearen Zusammenhang zwischen attentional load und Pupillengrösse.
- Die Personen stammen alle aus derselben Gruppe, aber unterschieden sich voneinander. Wir versuchen, den Mittelwert dieser Gruppe zu schätzen, und gleichzeitig, die Abweichung vom Mittelwert für jede Person.
Um die Interpretation eines Intercepts einfacher zu machen, zentrieren wir die Prädiktorvariable load, und speichern die zentrierte Variable als c_load. Der Intercept entspricht damit in unserem Regressionsmodell dem erwarteten Wert der abhängigen Variable bei einem mittleren attentional load (an der Stelle 0 von c_load).
pupilsize <- pupilsize %>%
mutate(c_load = load - mean(load))
Aufgabe 3
Schreiben Sie die Formel für dieses Modell auf. Sie wollen die erwartete Pupillengrösse als lineare Funktion des attentional load vorhersagen, mit “varying effects” für den Achsenabschnitt und den Koeffizienten von
c_load.Untersuchen Sie mit
get_prior()die Default Priors.
Die Formel, welche wir hier benutzen ist:
p_size ~ 1 + c_load + (1 + c_load | subj)
p_size ~ 1 + c_load + (1 + c_load | subj)Die fixed efects sind 1 + c_load, die varying effects der Versuchspersonen sind (1 + c_load | subj).
Die Prior Verteuliungen sind:
get_prior(p_size ~ 1 + c_load + (1 + c_load | subj), data = pupilsize)
prior class coef group resp dpar
(flat) b
(flat) b c_load
lkj(1) cor
lkj(1) cor subj
student_t(3, 5651.9, 2026.1) Intercept
student_t(3, 0, 2026.1) sd
student_t(3, 0, 2026.1) sd subj
student_t(3, 0, 2026.1) sd c_load subj
student_t(3, 0, 2026.1) sd Intercept subj
student_t(3, 0, 2026.1) sigma
nlpar bound source
default
(vectorized)
default
(vectorized)
default
default
(vectorized)
(vectorized)
(vectorized)
defaultWählen Sie für den Koeffizienten von c_load eine Normalverteilung mit Mittelwert 0 und einer Standardabweichung von 100. Dies bedeutet, dass wir mit 95% Sicherheit erwarten, dass der Effekt von c_load ungefähr im Bereich [-200, 200] liegt. Genauer können wir das so ausdrücken:
Aufgabe 4
Schätzen Sie das Modell mit Ihrer Formel und dem Prior für den Regressionskoeffizienten mit brms. Benutzen Sie das Argument
control = list(max_treedepth = 12), damit Sie von Stan keine Warnungen erhalten.Schauen Sie sich die
summary()an. Ist alles in Ordnung?Stellen Sie die Population-Level Effects grafisch dar (siehe Code weiter unten). Versuchen Sie, diese zu interpretieren.
summary(m1)
Family: gaussian
Links: mu = identity; sigma = identity
Formula: p_size ~ 1 + c_load + (1 + c_load | subj)
Data: pupilsize (Number of observations: 2228)
Samples: 4 chains, each with iter = 2000; warmup = 1000; thin = 1;
total post-warmup samples = 4000
Group-Level Effects:
~subj (Number of levels: 20)
Estimate Est.Error l-95% CI u-95% CI Rhat
sd(Intercept) 2639.73 433.59 1941.73 3614.58 1.01
sd(c_load) 70.47 14.66 47.09 103.64 1.01
cor(Intercept,c_load) 0.29 0.22 -0.16 0.68 1.00
Bulk_ESS Tail_ESS
sd(Intercept) 824 1677
sd(c_load) 1167 2001
cor(Intercept,c_load) 1310 1765
Population-Level Effects:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
Intercept 5651.47 586.12 4492.21 6835.70 1.01 478 742
c_load 60.59 16.71 27.78 93.51 1.00 1427 1820
Family Specific Parameters:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
sigma 505.28 7.59 490.58 520.40 1.00 5657 2572
Samples were drawn using sampling(NUTS). For each parameter, Bulk_ESS
and Tail_ESS are effective sample size measures, and Rhat is the potential
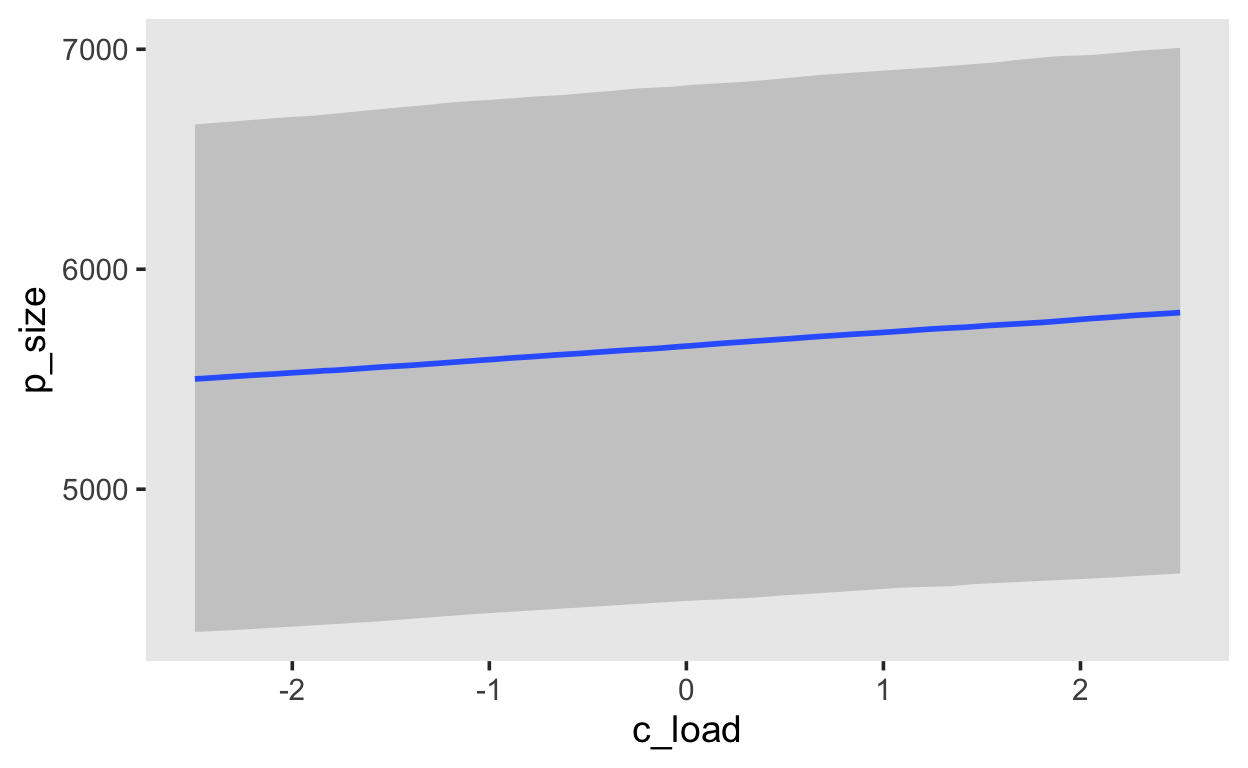
scale reduction factor on split chains (at convergence, Rhat = 1).Der Intercept repräsentiert die erwartete durschnittliche Pupillengrösse beim Wert 0 des zentrierten Prädiktors, welcher wiederum den Mittelwert des unzentrierten Prädiktor darstellt. Deshalb ist der erwartete Wert beim mittleren attentional load \(5651.47\) (95% Credible Interval: [4492.21, 6835.70]). Der Koeffizient c_load mit einem Mittelwert von \(60.59\) (95% Credible Interval: [27.78, 93.51]) ist der erwartete Zuwachs in per Pupillengrösse, wenn wir den attentional load um eine Einheit erhöhen.
Mit folgendem Code können Sie die Population-Level Effects darstellen.
library(patchwork)
p_intercept <- m1 %>%
mcmc_plot("b_Intercept", point_est = "mean", prob = 0.8, prob_outer = 0.95)
p_attentionalload <- m1 %>%
mcmc_plot("b_c_load", point_est = "mean", prob = 0.8, prob_outer = 0.95)
p_intercept / p_attentionalload

Mit der Funktion conditional_effects() können Sie den bedingten Efekt des Prädiktors auf die abhängige Variable darstellen, mit einem 95% Credible Interval.
conditional_effects(m1, prob = 0.95)
Aufgabe 5
Schätzen Sie mit der Savage-Dickey Methode einen Bayes Factor für die Hypothese, dass der Effekt von attentional load > 0 ist. Um diese Hypothese auszudrücken benutzen wir den Prior
prior(normal(0, 100), class = b, lb = 0). Das Argumentlb = 0heisst hier, der Parameter hat einen lower bound (Untergrenze) von 0. Dies führt dazu, dass alle Werte \(\leq 0\) unmöglich sind, da sie eine Wahrscheinlichkeitsdichte von 0 haben. Vergessen Sie nicht das Argumentsample_prior = TRUE, damit die Samples aus den Prior Verteilungen gespeichert werden.Berichten Sie den Bayes Factor (Wenn Sie den Ouput der
hypothesis()Funktion alsbfspeichern, dann ist der BF1/bf$hypothesis$Evid.Ratio).
bf <- hypothesis(m_savage_dickey, "c_load = 0")
1/bf$hypothesis$Evid.Ratio
[1] 86.34568Wir erhalten einen Bayes Factor \(BF_{10}\) für die Alternativhypothese dass der Effekt positiv ist. Die Daten sind unter dieser Hypothese ca. 86 mal wahrscheinlicher als unter der Nullhypothese.
Optionale Aufgabe
Versuchen Sie, mit der Funktion bayes_factor() einen Bayes Factor für die Hypothese, dass der Effekt von attentional load > 0 ist. Sie brauchen dafür zwei Modelle; eines mit dem Population-Level Effekt von c_load, das andere ohne diesen Effekt (aber sonst in jeder Hinsicht identisch).
Sie brauchen die zusätzlichen Argumente iter = 1e4, warmup = 1000 und save_pars = save_pars(all = TRUE). Mit dem ersteren erhalten Sie 10000 Samples aus dem Posterior, mit zweiterem sagen Sie, dass Sie 9000 Samples behalten wollen, und mit dem letzten Argument speichern Sie alle Parameter (diese werden für den bridge sampling Algorithmus gebraucht).
iterations <- 1e4
m_positive <- brm(p_size ~ 1 + c_load + (1 + c_load | subj),
prior = prior(normal(0, 100), class = b, lb = 0) +
prior(lkj(2), class = cor),
data = pupilsize,
iter = iterations, warmup = 1000,
cores = 4,
save_pars = save_pars(all = TRUE),
control = list(max_treedepth = 12),
file = "models/ex5-m_positive",
file_refit = "on_change")
Zusätzlich müssen wir das Nullmodell explizit definieren. Da uns der Fixed Effect von der zentrierten attentional load Variable interessiert, lassen wir diesen beim Nullmodel weg, lassen alles andere aber gleich. Da der Fixed Effect nicht mehr im Modell ist, brauchen wir auch den Prior normal(0, 100) nicht mehr.
Den Bayes Factor für unsere Alternativhypothese erhalten wir mit der Funktion bayes_factor(). Diese liefert uns den Bayes Factor für das erste Argument, in diesem Fall also m_positive.
BF <- bayes_factor(m_positive, m_null,
maxiter = 2e3,
silent = TRUE,
cores = 4)
BF
Estimated Bayes factor in favor of m_positive over m_null: 122.49819Wir erhalten hier einen Bayes Factor von ca. 122.5. Dies bedeutet, dass die Daten unter der Hypothese, dass der lineare Effekt von attentional positiv ist, rund 122 mal wahrscheinlicher als unter der Nullhypothese sind.
Sowohl der erste Bayes Factor (Savage-Dickey) als der zweite (bridge sampling) sind relativ gross (“strong” bis “decisive”), und demonstrieren Evidenz gegen die Nullhypothese. Die Werte der erhaltenen Bayes Factors sind ziemlich variabel, obschon sie relativ zuverlässig Evidenz für die Alternativhypothese und gegen die Nullhypothese liefern. Generell ist die bridge sampling Methode etwas robuster, aber es wäre hier angebracht, die Modelle mehrere Male zu schätzen, und dann die resultierenden Bayes Factors zu ermitteln, um die Variabilität berichten zu können.