Parameterschätzung mit Linearen Modellen
Mittelwert und Standardabweichung einer Normalverteilung schätzen
Wir haben 3 Datenpunkte, von denen wir annehmen können, dass sie aus einer Normalverteilung kommen. Die drei Werte sind \(y_1 = 85\), \(y_2 = 100\), und \(y_3 = 115\). Die Wahrscheinlichkeit eines Datenpunktes is gegeben durch
\[ p(y | \mu, \sigma) = \frac{1}{Z} exp \left(- \frac{1}{2} \frac{(y-\mu)^2}{\sigma^2}\right) \]
\[ Z = \sigma \sqrt{2\pi} \] ist eine Normalisierungskonstante.
Damit können wir die Wahrscheinlichkeit eines Datenpunktes berechnen, wenn wir die Parameter \(\mu\) und \(\sigma\) kennen. Unter der Annahme, dass die drei Datenpunkte unabhängig sind, ist die Wahrscheinlichkeit der Daten das Produkt der Einzelwahrscheinlichkeiten.
Wie aber finden wir die beiden Parameter der Normalverteilung, \(\mu\) und \(\sigma\)?
Graphical model
Wir können wir schon im letzten Kapitel das Problem als Graphical Model darstellen. Dies hilft, das Problm zu veranschaulichen; vor allem wenn wir komplexere Probleme betrachten, kann dies hilfreich sein.
Die Variablen in diesem Modell sind:
\[ \mu \sim ??? \] \[ \sigma \sim ??? \] \[ y \sim N(\mu, \sigma) \]
\(y\) ist eine beobachtete Variable, und \(\mu\) und \(\sigma\) sind parameter, welche geschätzt werden müssen. Dies können wir mit der Maximum-Likelihood Methode machen, um Punktschätzungen zu erhalten. Der folgende Code illustriert, wie das ungefähr funktioniert. Wir suche eine Kombination von \(\mu\) und \(\sigma\), für welceh die Wahrscheinlichkeit am grössten ist, genau diese drei Dantepunkte zu beobachten. Anstatt alle möglichen Werte zu betrachten, wie in der Maximum-Likelihood Methode, nehmen wir drei Beispielswerte für \(\mu\), \(87.8, 100\) und \(112\), und drei Werte für \(\sigma\), \(7.35, 12.2\) und \(18.4\). Wir erstellen dann die 9 möglichen Kombinationen dieser Werte, und benutzten ein Grid (von 50 bis 150) für die möglichen Datenpunkte.
sequence_length <- 100
d1 <- crossing(y = seq(from = 50, to = 150, length.out = sequence_length),
mu = c(87.8, 100, 112),
sigma = c(7.35, 12.2, 18.4)) %>%
mutate(density = dnorm(y, mean = mu, sd = sigma),
mu = factor(mu, labels = str_c("mu==", c(87.8, 100, 112))),
sigma = factor(sigma,
labels = str_c("sigma==", c(7.35, 12.2, 18.4))))
theme_set(
theme_bw() +
theme(text = element_text(color = "white"),
axis.text = element_text(color = "black"),
axis.ticks = element_line(color = "white"),
legend.background = element_blank(),
legend.box.background = element_rect(fill = "white",
color = "transparent"),
legend.key = element_rect(fill = "white",
color = "transparent"),
legend.text = element_text(color = "black"),
legend.title = element_text(color = "black"),
panel.background = element_rect(fill = "white",
color = "white"),
panel.grid = element_blank()))
In Grafik 2 werden nun die 9 Kombinationen, und die 3 beobachteten Datenpunkte (in Rot) dargestellt. Die Paratemeter \(\mu\) und \(\sigma\) definieren jeweils eine Normalverteilung, und die gestrichelte Linie zeigt die Wahrscheinlichkeit eines Datenpunktes unter dieser Verteilung. Die Wahrscheinlichkeit der Daten ist gegeben durch das Produkt der Wahrscheinlichkeiten. Diejenigen Paramterwerte, welche die Wahrscheinlichkeit maximieren, “gewinnen.” In diesem Beispiel wäre das die mittlere Verteilung.
d1 %>%
ggplot(aes(x = y)) +
geom_ribbon(aes(ymin = 0, ymax = density),
fill = "steelblue") +
geom_vline(xintercept = c(85, 100, 115),
linetype = 3, color = "white") +
geom_point(data = tibble(y = c(85, 100, 115)),
aes(y = 0.002),
size = 2, color = "red") +
scale_y_continuous(expression(italic(p)(italic(y)*"|"*mu*", "*sigma)),
expand = expansion(mult = c(0, 0.05)), breaks = NULL) +
ggtitle("Welche Normalverteilung?") +
coord_cartesian(xlim = c(60, 140)) +
facet_grid(sigma ~ mu, labeller = label_parsed) +
theme_bw() +
theme(panel.grid = element_blank())
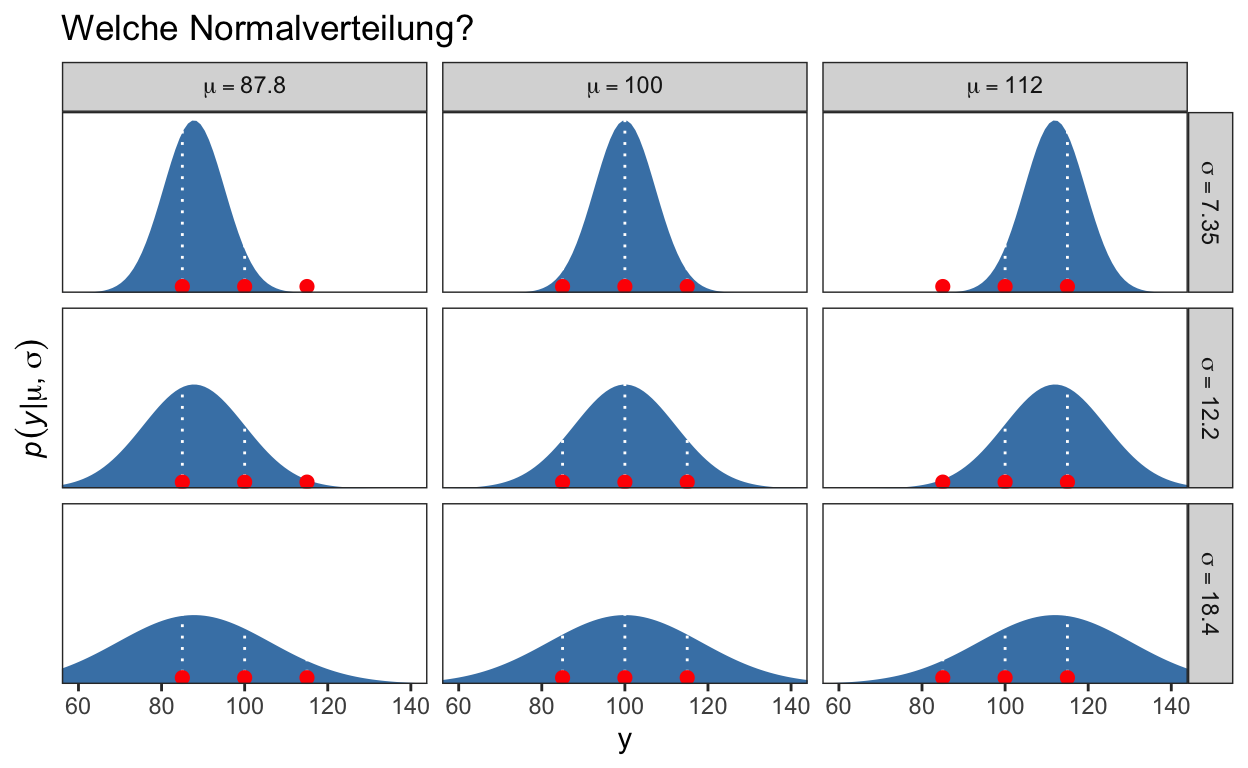
Figure 2: Kombinationen von \(\mu\) und \(\sigma\) Parameterwerten.
Beispiel: t-Test
Um die Parameter einer Normalverteilung mittels Markov Chain Monte Carlo Sampling Bayesian zu schätzen, nehmen wir ein Beispiel aus dem Buch Doing Bayesian Data Analysis (Kruschke 2015).
Wir haben IQ Scores einer Stichprobe, welche eine “Smart Drug” konsumiert hat. IQ Werte sind so normiert, dass wir in der Bevölkerung einen Mittelwert von 100, und eine SD von 15 haben. Nun wollen wir wissen, ob und wie sich die “Smart Drug” Gruppe von von diesem erwarteten Mittelwert unterscheidet. Gleichzeitig haben wir auch eine Gruppe, welche ein Placebo konsumiert hat.
Wir können die Daten von Hand eingeben, und zu einem Dataframe zusammenfügen.
smart = tibble(IQ = c(101,100,102,104,102,97,105,105,98,101,100,123,105,103,
100,95,102,106,109,102,82,102,100,102,102,101,102,102,
103,103,97,97,103,101,97,104,96,103,124,101,101,100,
101,101,104,100,101),
Group = "SmartDrug")
placebo = tibble(IQ = c(99,101,100,101,102,100,97,101,104,101,102,102,100,105,
88,101,100,104,100,100,100,101,102,103,97,101,101,100,101,
99,101,100,100,101,100,99,101,100,102,99,100,99),
Group = "Placebo")
TwoGroupIQ <- bind_rows(smart, placebo) %>%
mutate(Group = fct_relevel(as.factor(Group), "Placebo"))
Mit diesen Daten könnten wir einen t-Test machen, um die Mittelwerte der beiden Gruppen zu vergleichen. Die Mittelwertem sowie die beiden Standardabweichungen, können wir so schätzen.
library(kableExtra)
TwoGroupIQ %>%
group_by(Group) %>%
summarise(mean = mean(IQ),
sd = sd(IQ)) %>%
mutate(across(where(is.numeric), round, 2)) %>%
kbl() %>%
kable_styling(bootstrap_options = c("striped", "hover"))
| Group | mean | sd |
|---|---|---|
| Placebo | 100.36 | 2.52 |
| SmartDrug | 101.91 | 6.02 |
Die Smart Drug Gruppe hat einen leicht grösseren Mittelwert, aber auch eine grössere Standardabweichung, weshlab wir hier einen Welch-Test einem t-Test vorziehen würden.
t.test(IQ ~ Group,
data = TwoGroupIQ)
Welch Two Sample t-test
data: IQ by Group
t = -1.6222, df = 63.039, p-value = 0.1098
alternative hypothesis: true difference in means between group Placebo and group SmartDrug is not equal to 0
95 percent confidence interval:
-3.4766863 0.3611848
sample estimates:
mean in group Placebo mean in group SmartDrug
100.3571 101.9149 Mit diesem nicht-signifikanten Welch-Test gibt es nun nicht viele Möglichkeiten, weiterzufahren. Slebstverständlich hätten wir die Power dieses Test erhöhen können, aber nachdem die Daten gesammelt wurden, ist diese Erkenntnis nicht sehr hilfreich.
Smart Drug Gruppe
Wir ignorieren zunächst die Kontrollgruppe, und versuchen, die Parameter der Smart Drug Gruppe mit dem brms Package zu schätzen.
Wir erstelln zuerst einen Subdatensatz:
d <- TwoGroupIQ %>%
filter(Group == "SmartDrug") %>%
mutate(Group = fct_drop(Group))
d %>%
ggplot(aes(x = IQ)) +
geom_histogram(fill = "skyblue3", binwidth = 1) +
scale_y_continuous(expand = expansion(mult = c(0, 0.05))) +
theme_tidybayes()

Parameter schätzen mit brms
Wir wollen nun einen Mittelwert, und eine Standardabweichung mit brms schätzen. Bevor wir anfangen, überlegen wir uns zuerst, welche Prior Verteilungen denn überhaupt in Frage kommen. Für dieses Modell kennen wir bereits die Parameter, die wir schätzen müssen, aber in späteren Kapiteln werden wir es mir komplexeren Modellen zu tun haben, bei denen es nicht so offensichtilich ist.
Priors
Glücklicherweise bietet brms uns hier sehr viel Hilfe. Für die meisten Fälle können wir uns sogar darauf verlassen, dass die Default Priors genügen, so dass wir nicht einmal selber Priors definieren müssten. Es ist aber besser, wenn wir lernen, selber Prior Verteilungen zu definieren. In brms gibt es eine Funktion, get_prior(), mit welcher wir herausfinden können, welche Parameter Prior Verteilungen brauchen, und was die Defaults sind.
Diese Funktion nimmt als Argumente eine Formel, welche ein Regressionmodell spezifiert, und einen Dataframe. Alle weiteren Argumente sind optional. INdiesem Fall lautet unser Formel IQ ~ 1, was soviel bedeutet, wie: wir sagen den Erwartungswert vom IQ voraus, mit einem Achsenabschnitt (Intercept). Dieser Achsenabschnitt entspricht einem Parameter, den wir schätzen, und dieder Parameter wiederum entspricht dem Mittelwert \(\mu\). Der andere Parameter, \(\sigma\), wird hier nicht explizit erwähnt—dies ist ein sogennanter nuisance Parameter; disen müssen wir schätzen, interessieren uns aber nicht besonders dafür.
priors <- get_prior(IQ ~ 1,
data = d)
priors
prior class coef group resp dpar nlpar bound
student_t(3, 102, 3) Intercept
student_t(3, 0, 3) sigma
source
default
defaultStandardabweichung
Für den Parameter \(\sigma\) erhalten wir folgenden Vorschlag: eine student_t(3, 0, 3) Verteilung. Die ist ein t-Verteilung mit drei Parametern. Die Parameter zwei und drei mit den Werten 0 und 3 sind die beiden location und scale Parameter, ähnlich dem Mittelwert und der Standardabweichung einer Normalverteilung. Der erste Parameter, \(\nu\), hat hier den Wert 3. Dieser Parameter wird oft als Normalitätsparameter bezeichnet—wenn \(\nu \to \infty\) geht die t-Verteilung in eine Normalverteilung über. Grafik 3 zeigt diese t-Verteilung. Sie drückt aus, dass wir Werte zwischen 0 und ca. 4 für die Standardabweichung erwarten.
Was hier verschwiegen wird, ist dass Stan nur die positive Hälfte der t-Verteilung als Prior nimmt, denn der Parameter \(\sigma\) muss positiv sein. Folglich muss der Wertebereich der Verteilung \((0, \infty]\) sein. Wir haben es hier also mit einer halben t-Verteilung zu tun.
tibble(x = seq(from = 0, to = 10, by = .025)) %>%
mutate(d = dt(x, df = 3)) %>%
ggplot(aes(x = x, ymin = 0, ymax = d)) +
geom_ribbon(fill = "skyblue3") +
scale_x_continuous(expand = expansion(mult = c(0, 0.05))) +
scale_y_continuous(expand = expansion(mult = c(0, 0.05)), breaks = NULL) +
coord_cartesian(xlim = c(0, 8),
ylim = c(0, 0.35)) +
xlab(expression(sigma)) +
labs(subtitle = "Half-student-t Distribution: Prior für Standardabweichung.") +
theme_bw(base_size = 14)
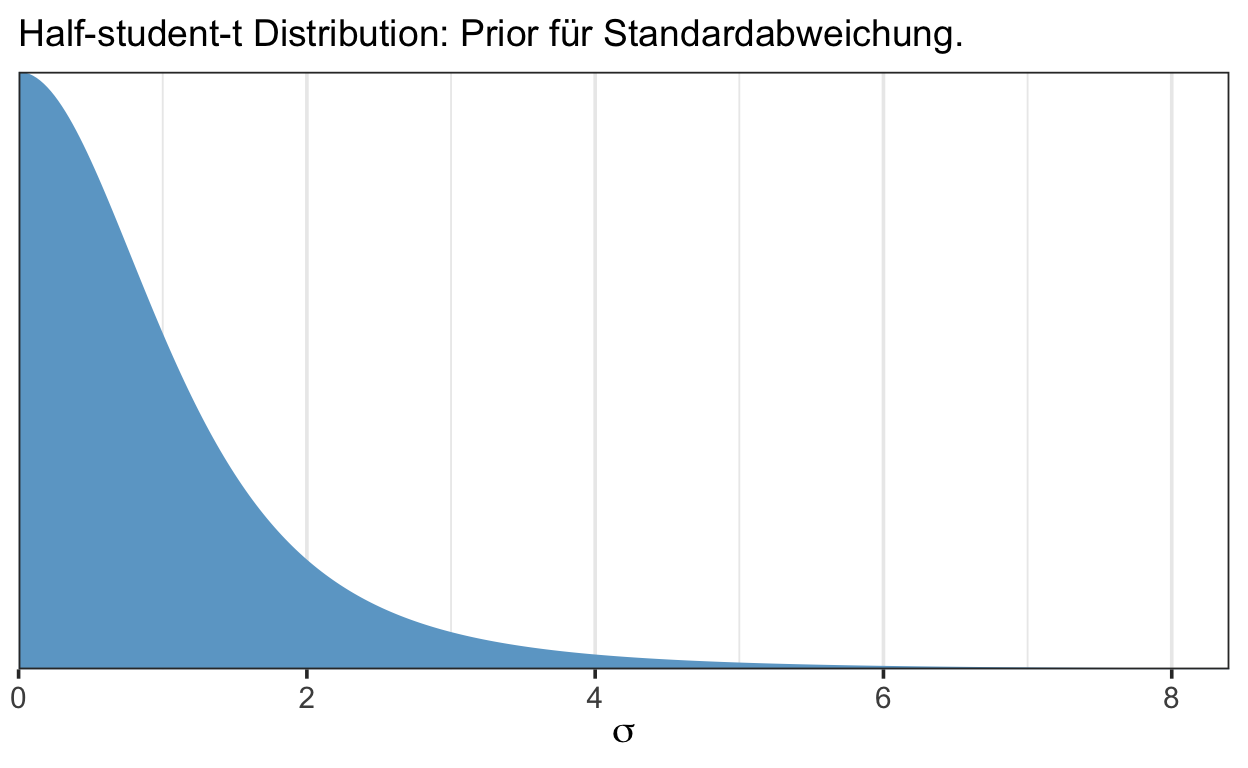
Figure 3: Prior für \(\sigma\).
Mittelwert
Für \(\mu\) erhalten wir ebenfalls eine t-Verteilung, student_t(3, 102, 3). Diese hat ebenfalls einen Normalitätsparameter \(\nu\) von 3, aber setzt die Location und Scale Parameter bei 102 und 3 (102 entspricht ungefähr dem Mittelwert). Grafik 4 yiegt diese Verteilung.
tibble(x = seq(from = 0, to = 200, by = .025)) %>%
mutate(d = dnorm(x, mean = 102, sd = 3)) %>%
ggplot(aes(x = x, ymin = 0, ymax = d)) +
geom_ribbon(fill = "skyblue3") +
scale_x_continuous(expand = expansion(mult = c(0, 0.05))) +
scale_y_continuous(expand = expansion(mult = c(0, 0.05)), breaks = NULL) +
coord_cartesian(xlim = c(50, 150),
ylim = c(0, 0.15)) +
xlab(expression(mu)) +
labs(subtitle = "Normalverteilter Prior für Mittelwert") +
theme_bw(base_size = 14)
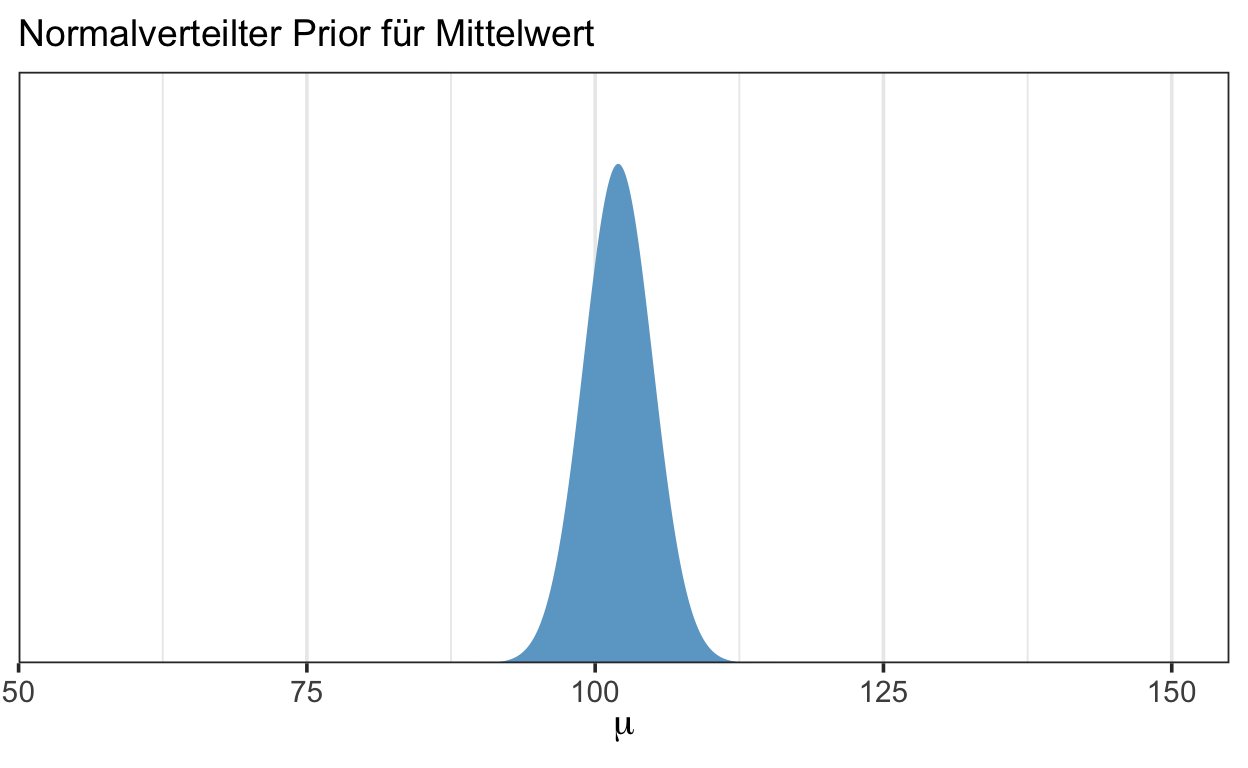
Figure 4: Prior für \(\mu\).
Prior Predictive Distribution
m1_prior <- brm(IQ ~ 1,
prior = priors,
data = d,
sample_prior = "only",
file = "models/twogroupiq-prior-1")
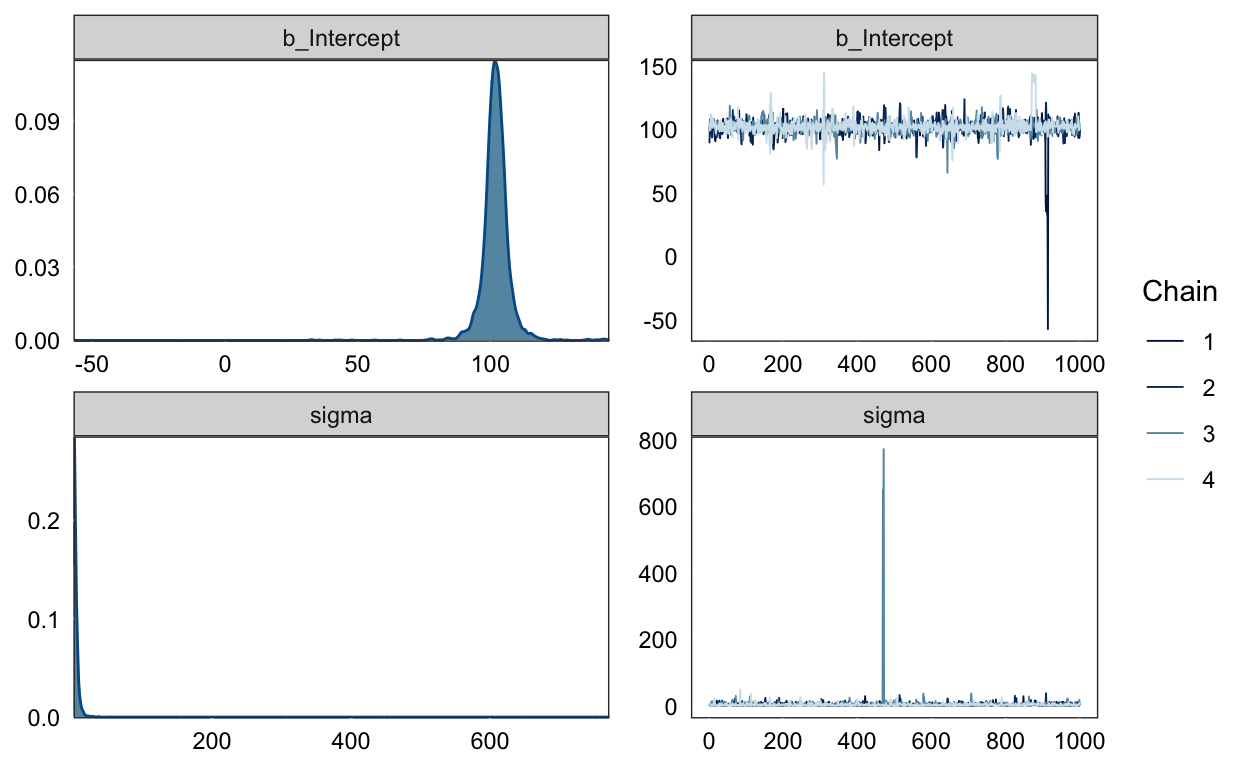
summary(m1_prior)
Family: gaussian
Links: mu = identity; sigma = identity
Formula: IQ ~ 1
Data: d (Number of observations: 47)
Samples: 4 chains, each with iter = 2000; warmup = 1000; thin = 1;
total post-warmup samples = 4000
Population-Level Effects:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
Intercept 101.95 6.20 92.34 111.71 1.00 2048 1510
Family Specific Parameters:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
sigma 3.75 17.71 0.10 12.94 1.00 1757 1199
Samples were drawn using sampling(NUTS). For each parameter, Bulk_ESS
and Tail_ESS are effective sample size measures, and Rhat is the potential
scale reduction factor on split chains (at convergence, Rhat = 1).plot(m1_prior)
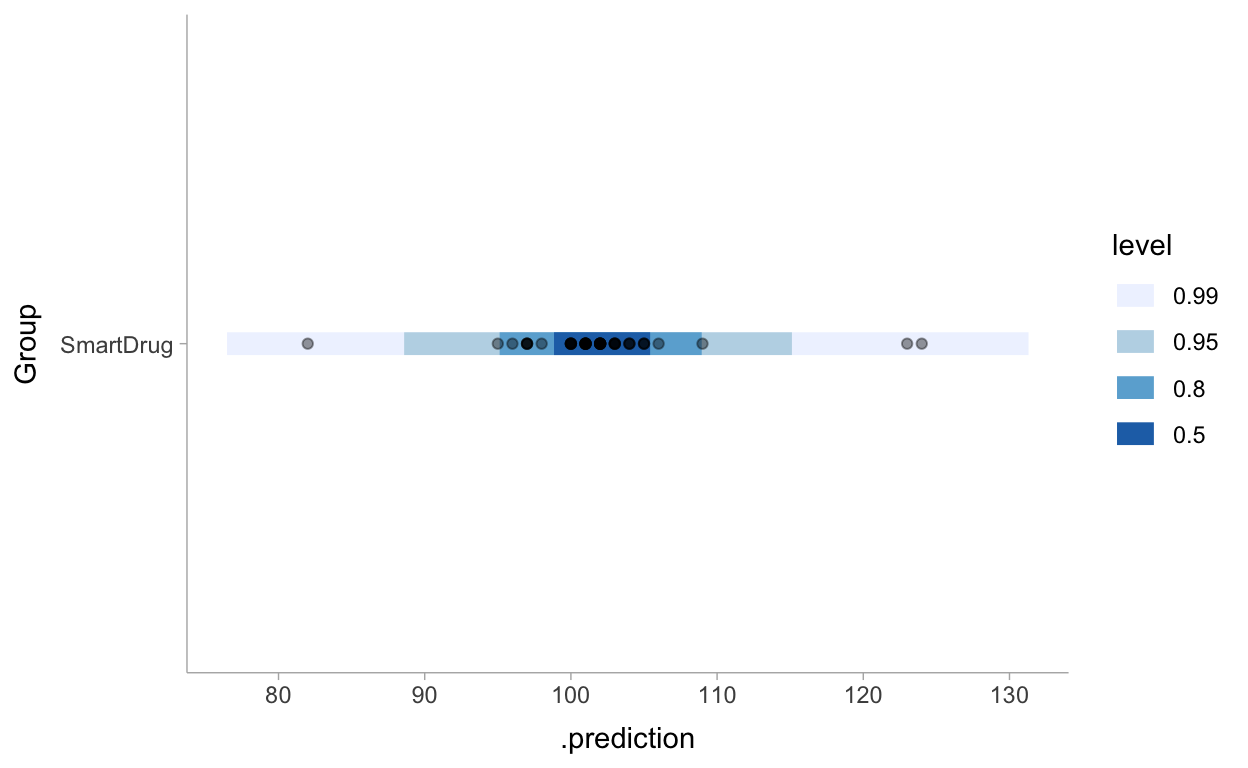
library(tidybayes)
prior_pred_1 <- d %>%
modelr::data_grid(Group) %>%
add_predicted_draws(m1_prior) %>%
ggplot(aes(y = Group, x = .prediction)) +
stat_interval(.width = c(.50, .80, .95, .99)) +
geom_point(aes(x = IQ), alpha = 0.4, data = d) +
scale_color_brewer() +
theme_tidybayes()
prior_pred_1
Die Posterior Verteilung durch Sampling approximieren
m1 <- brm(IQ ~ 1,
prior = priors,
data = d,
file = "models/twogroupiq-1")
plot(m1)
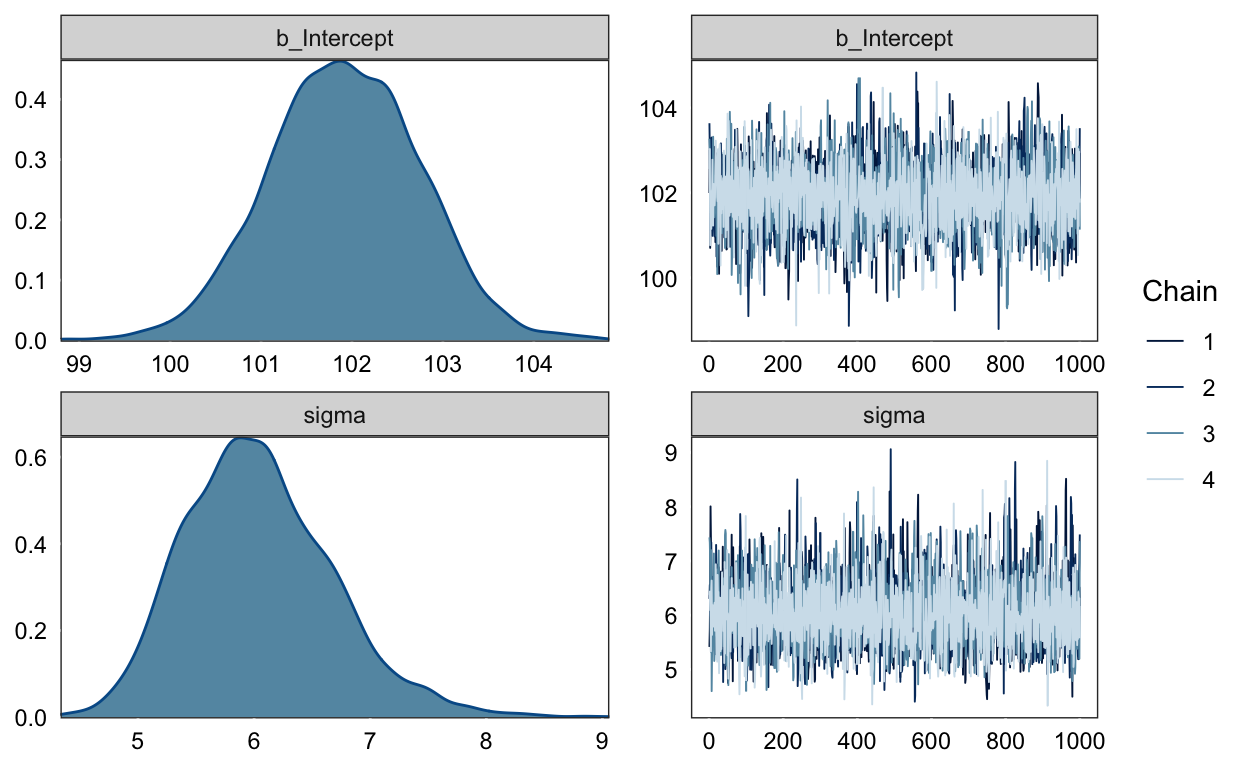
print(m1)
Family: gaussian
Links: mu = identity; sigma = identity
Formula: IQ ~ 1
Data: d (Number of observations: 47)
Samples: 4 chains, each with iter = 2000; warmup = 1000; thin = 1;
total post-warmup samples = 4000
Population-Level Effects:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
Intercept 101.92 0.83 100.31 103.53 1.00 3134 2075
Family Specific Parameters:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
sigma 6.04 0.64 4.96 7.47 1.00 3060 2458
Samples were drawn using sampling(NUTS). For each parameter, Bulk_ESS
and Tail_ESS are effective sample size measures, and Rhat is the potential
scale reduction factor on split chains (at convergence, Rhat = 1).Posterior Quantile Intervals
mcmc_plot(m1, pars = "b_")
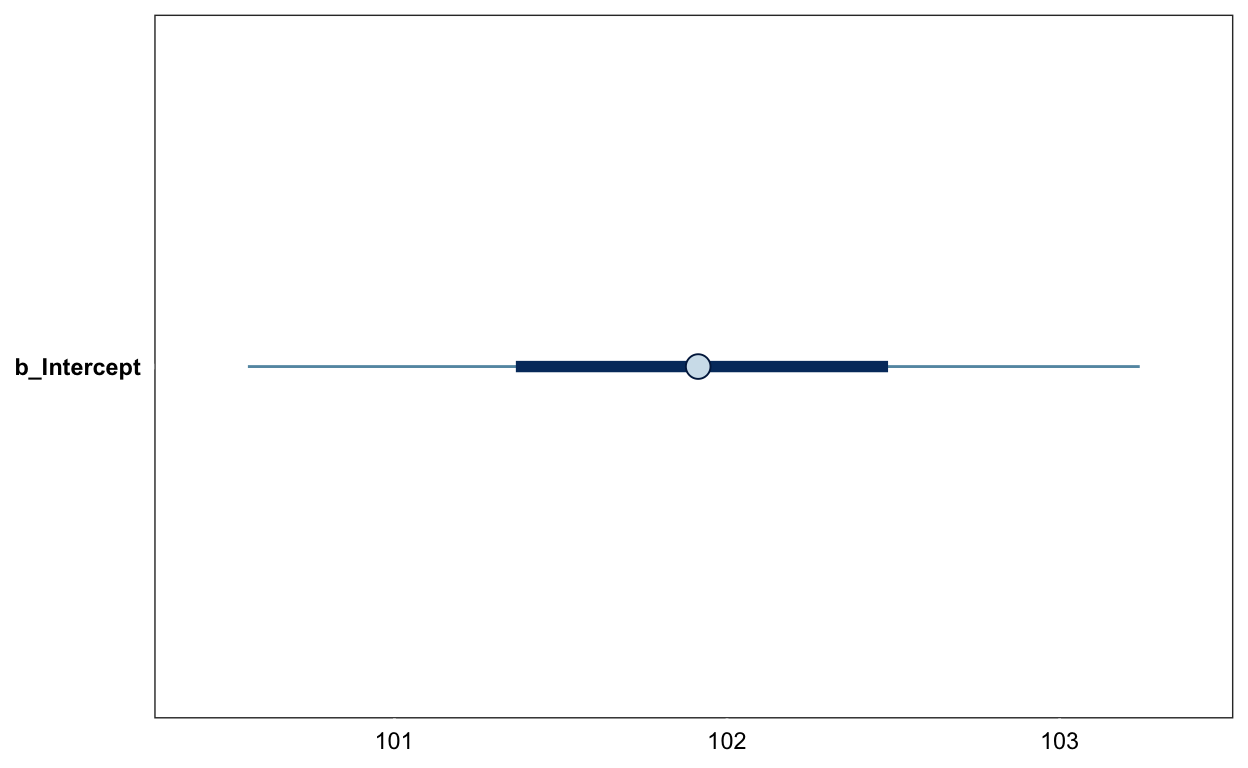
mcmc_plot(m1, pars = "sigma")
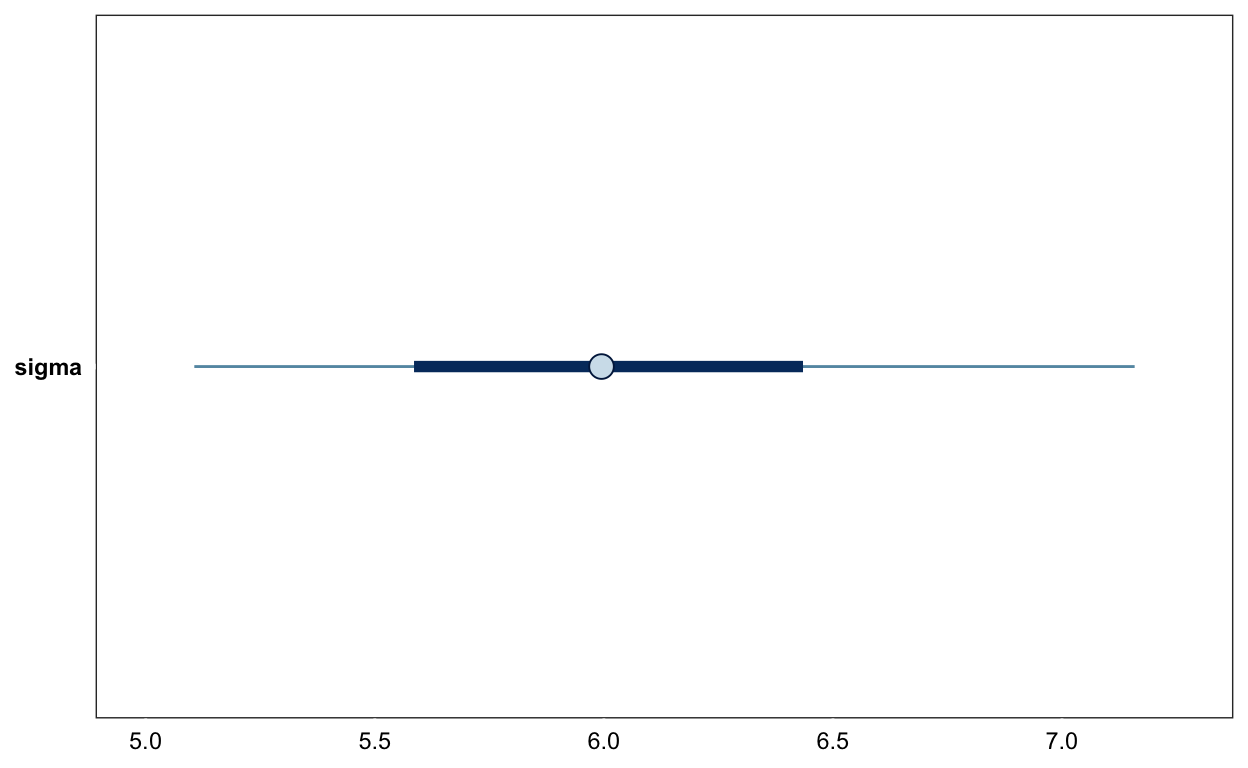
Posterior Samples extrahieren
samples <- posterior_samples(m1) %>%
transmute(mu = b_Intercept, sigma = sigma)
mu .lower .upper .width .point .interval
1 101.9132 101.36505 102.4841 0.50 median qi
2 101.9132 100.86039 102.9730 0.80 median qi
3 101.9132 100.30502 103.5255 0.95 median qi
4 101.9132 99.71696 104.1686 0.99 median qisamples %>%
select(mu) %>%
median_qi(.width = c(.50, .80, .95, .99)) %>%
ggplot(aes(x = mu, xmin = .lower, xmax = .upper)) +
geom_pointinterval() +
ylab("") +
theme_tidybayes()
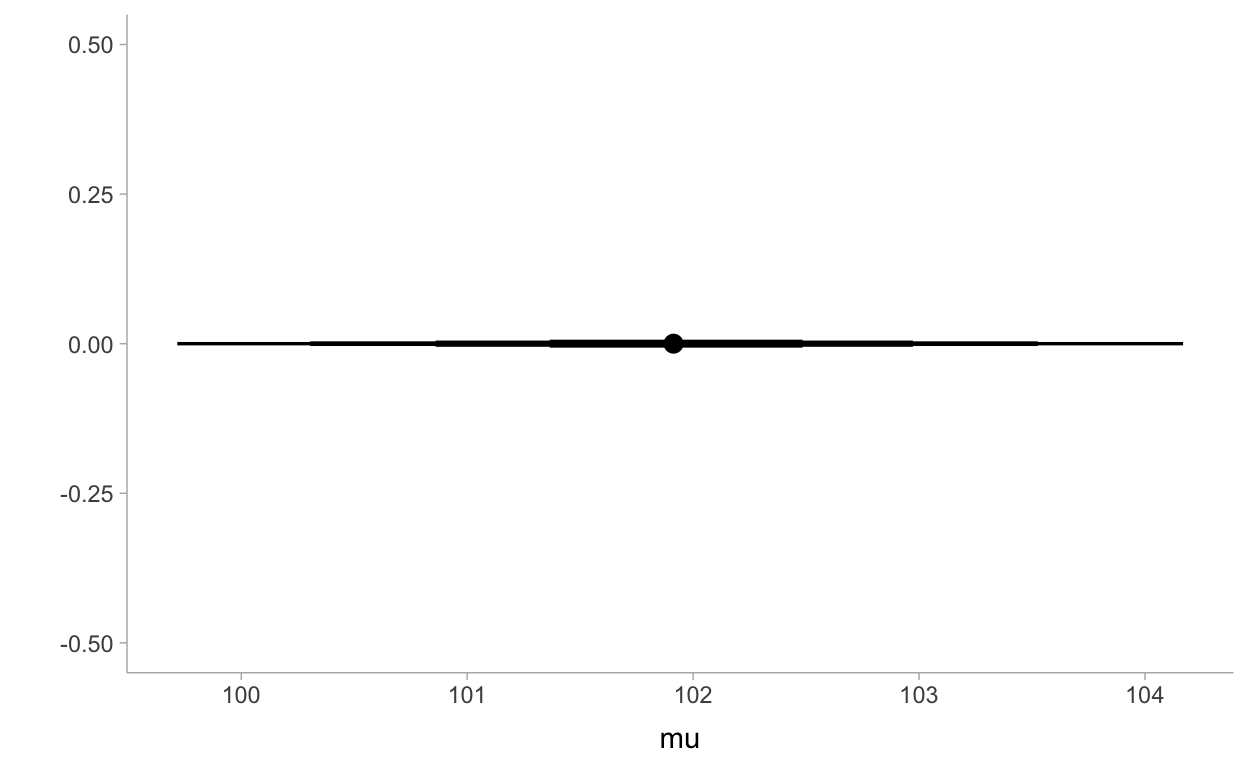
samples %>%
select(mu) %>%
ggplot(aes(x = mu)) +
stat_halfeye() +
theme_tidybayes()

samples %>%
select(sigma) %>%
ggplot(aes(x = sigma)) +
stat_halfeye(point_interval = mode_hdi) +
theme_tidybayes()
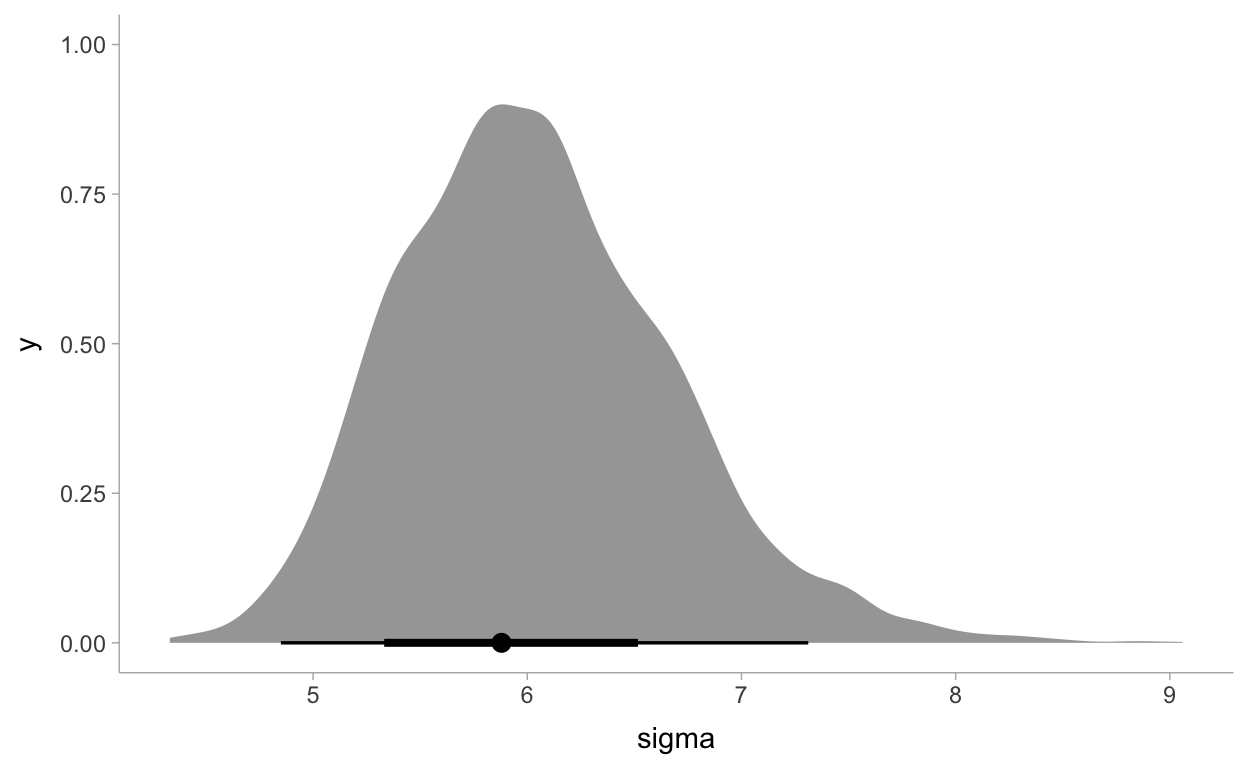
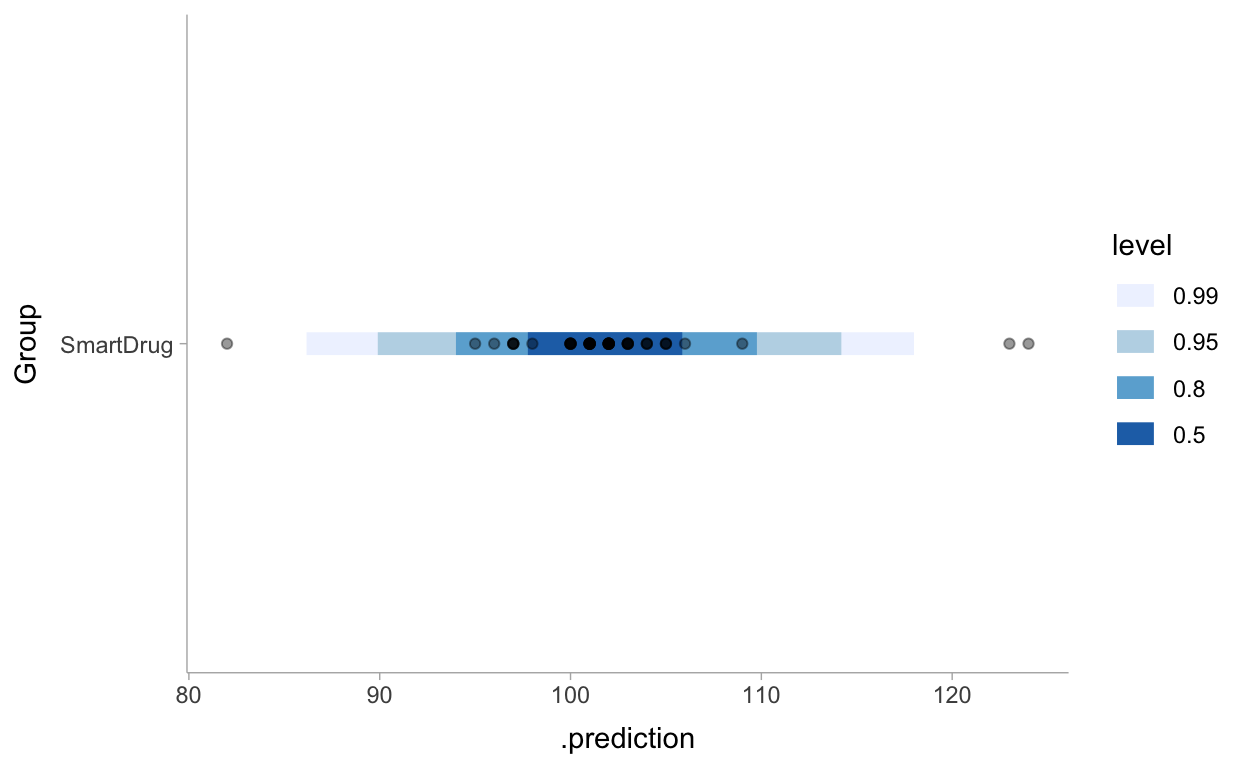
Posterior Predictive Distribution
post_pred_1 <- d %>%
modelr::data_grid(Group) %>%
add_predicted_draws(m1) %>%
ggplot(aes(y = Group, x = .prediction)) +
stat_interval(.width = c(.50, .80, .95, .99)) +
geom_point(aes(x = IQ), alpha = 0.4, data = d) +
scale_color_brewer() +
theme_tidybayes()
post_pred_1
Prior vs Posterior Predictive Distribution
# cowplot fall nötig installieren
if (!("cowplot" %in% installed.packages())) {install.packages("cowplot")}
cowplot::plot_grid(prior_pred_1,
post_pred_1,
labels = c('Prior predictive', 'Posterior predictive'),
label_size = 12,
align = "h",
nrow = 2)

Zwei Gruppen
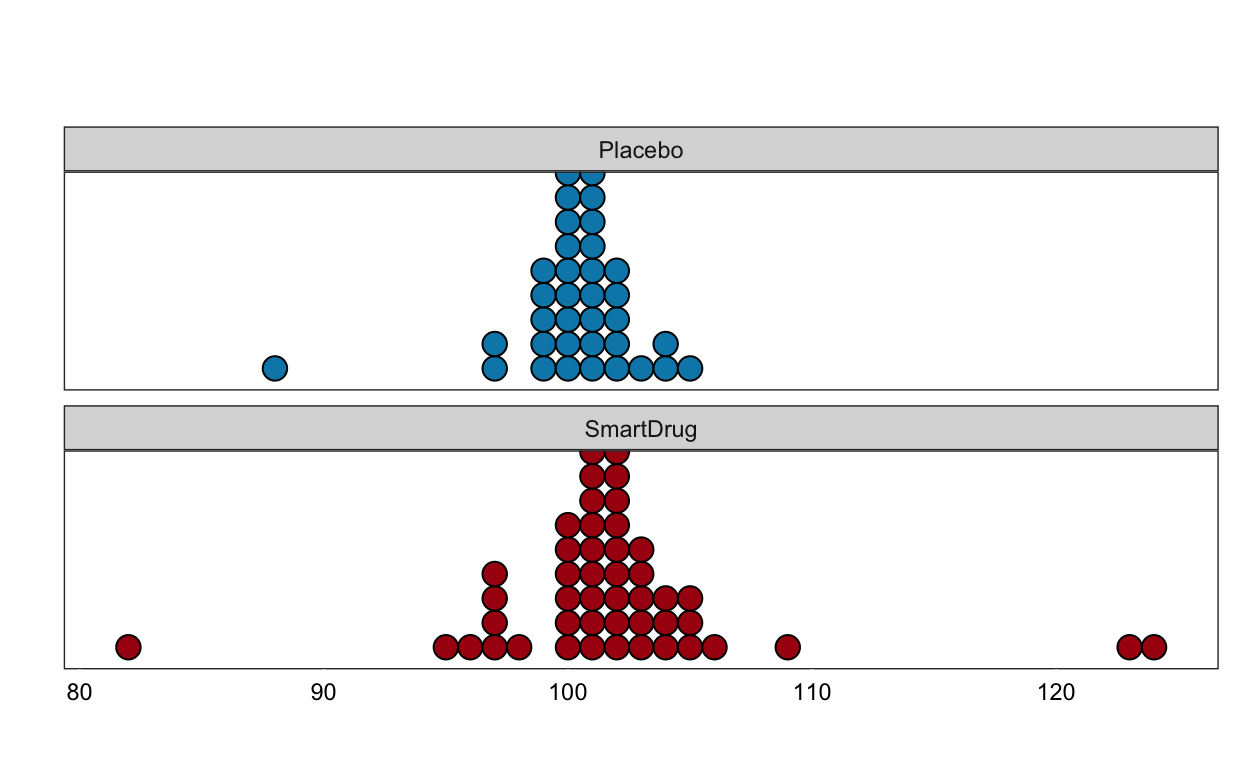
TwoGroupIQ %>%
ggplot(aes(x = IQ, fill = Group)) +
geom_dotplot(binwidth = 1) +
scale_fill_manual(values = c("#0288b7", "#a90010"), guide = FALSE) +
scale_y_continuous(breaks = NULL) +
labs(y = "Count", x = "IQ") +
facet_wrap(~ Group, nrow = 2) +
plot_annotation(title = "IQ difference",
subtitle = "Smart drug vs placebo",
theme = theme(plot.title = element_text(face = "bold",
size = rel(1.5))))
t-Test als Allgemeines Lineares Modell
Wir nehmen hier zunächst an, dass die beiden Gruppen dieselbe Varainz haben (Varianzgleichheit), obwohl wir vermuten, dass dies nicht der Fall ist.
Tradionelle Notation
\[y_{ij} = \alpha + \beta x_{ij} + \epsilon_{ij}\]
\[\epsilon \sim N(0, \sigma^2)\]
Probabilistische Notation
\[y_{ij} \sim N(\mu, \sigma^2)\] \[\mu_{ij} = \alpha + \beta x_{ij}\]
\(X_{ij}\) ist eine Indikatorvariable.
Ordinary Least Squares Schätzung
levels(TwoGroupIQ$Group)
[1] "Placebo" "SmartDrug"Mit R Formelnotation:
fit_ols <- lm(IQ ~ Group,
data = TwoGroupIQ)
summary(fit_ols)
Call:
lm(formula = IQ ~ Group, data = TwoGroupIQ)
Residuals:
Min 1Q Median 3Q Max
-19.9149 -0.9149 0.0851 1.0851 22.0851
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 100.3571 0.7263 138.184 <2e-16 ***
GroupSmartDrug 1.5578 0.9994 1.559 0.123
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 4.707 on 87 degrees of freedom
Multiple R-squared: 0.02717, Adjusted R-squared: 0.01599
F-statistic: 2.43 on 1 and 87 DF, p-value: 0.1227Dummy Coding in R
contrasts(TwoGroupIQ$Group)
SmartDrug
Placebo 0
SmartDrug 1mm1 <- model.matrix(~ Group, data = TwoGroupIQ)
head(mm1)
(Intercept) GroupSmartDrug
1 1 1
2 1 1
3 1 1
4 1 1
5 1 1
6 1 1as_tibble(mm1) %>%
group_by(GroupSmartDrug) %>%
slice_sample(n= 3)
# A tibble: 6 x 2
# Groups: GroupSmartDrug [2]
`(Intercept)` GroupSmartDrug
<dbl> <dbl>
1 1 0
2 1 0
3 1 0
4 1 1
5 1 1
6 1 1Dummy Coding in R
as_tibble(mm1) %>%
group_by(GroupSmartDrug) %>%
slice_sample(n= 3)
# A tibble: 6 x 2
# Groups: GroupSmartDrug [2]
`(Intercept)` GroupSmartDrug
<dbl> <dbl>
1 1 0
2 1 0
3 1 0
4 1 1
5 1 1
6 1 1mm2 <- model.matrix(~ 0 + Group, data = TwoGroupIQ)
as_tibble(mm2) %>%
group_by(GroupSmartDrug) %>%
slice_sample(n= 3)
# A tibble: 6 x 2
# Groups: GroupSmartDrug [2]
GroupPlacebo GroupSmartDrug
<dbl> <dbl>
1 1 0
2 1 0
3 1 0
4 0 1
5 0 1
6 0 1Graphical Model
Get Priors
priors2 <- get_prior(IQ ~ 1 + Group,
data = TwoGroupIQ)
priors2
prior class coef group resp dpar
(flat) b
(flat) b GroupSmartDrug
student_t(3, 101, 2.5) Intercept
student_t(3, 0, 2.5) sigma
nlpar bound source
default
(vectorized)
default
defaultGet Priors
priors3 <- get_prior(IQ ~ 0 + Group,
data = TwoGroupIQ)
priors3
prior class coef group resp dpar nlpar
(flat) b
(flat) b GroupPlacebo
(flat) b GroupSmartDrug
student_t(3, 0, 2.5) sigma
bound source
default
(vectorized)
(vectorized)
defaultPriors definieren und vom Prior Sampeln
priors2_b <- prior(normal(0, 2), class = b)
m2_prior <- brm(IQ ~ 1 + Group,
prior = priors2_b,
data = TwoGroupIQ,
sample_prior = "only",
file = "models/twogroupiq-prior-2")
prior_pred_2 <- TwoGroupIQ %>%
modelr::data_grid(Group) %>%
add_predicted_draws(m2_prior) %>%
ggplot(aes(y = Group, x = .prediction)) +
stat_interval(.width = c(.50, .80, .95, .99)) +
geom_point(aes(x = IQ), alpha = 0.4, data = TwoGroupIQ) +
scale_color_brewer() +
theme_tidybayes()

Priors definieren und vom Prior Sampeln
priors3_b <- prior(normal(100, 10), class = b)
m3_prior <- brm(IQ ~ 0 + Group,
prior = priors3_b,
data = TwoGroupIQ,
sample_prior = "only",
file = "models/twogroupiq-prior-3")
prior_pred_3 <- TwoGroupIQ %>%
modelr::data_grid(Group) %>%
add_predicted_draws(m3_prior) %>%
ggplot(aes(y = Group, x = .prediction)) +
stat_interval(.width = c(.50, .80, .95, .99)) +
geom_point(aes(x = IQ), alpha = 0.4, data = TwoGroupIQ) +
scale_color_brewer() +
theme_tidybayes()
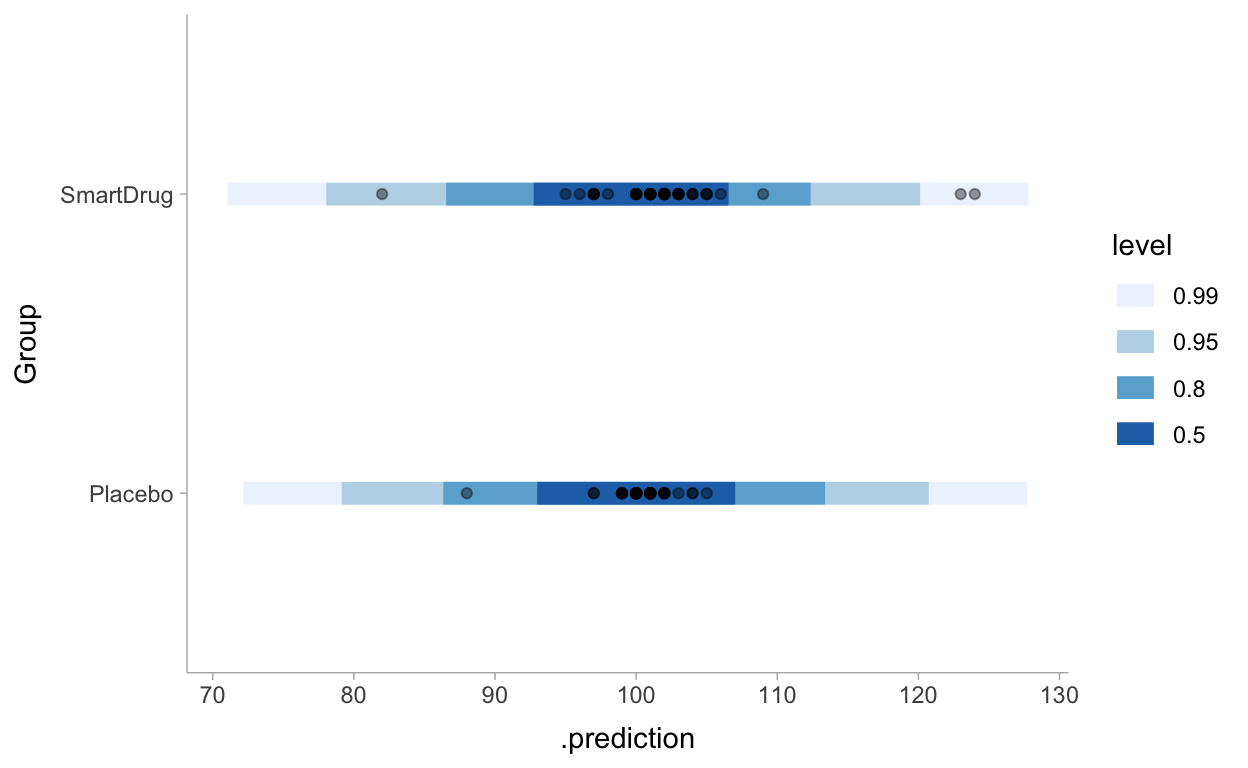
Posterior Sampling
m2 <- brm(IQ ~ 1 + Group,
prior = priors2_b,
data = TwoGroupIQ,
file = "models/twogroupiq-2")
m3 <- brm(IQ ~ 0 + Group,
prior = priors3_b,
data = TwoGroupIQ,
file = "models/twogroupiq-3")
Summary
summary(m2)
Family: gaussian
Links: mu = identity; sigma = identity
Formula: IQ ~ 1 + Group
Data: TwoGroupIQ (Number of observations: 89)
Samples: 4 chains, each with iter = 2000; warmup = 1000; thin = 1;
total post-warmup samples = 4000
Population-Level Effects:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS
Intercept 100.51 0.69 99.18 101.90 1.00 3735
GroupSmartDrug 1.23 0.88 -0.49 2.95 1.00 3633
Tail_ESS
Intercept 2921
GroupSmartDrug 3160
Family Specific Parameters:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
sigma 4.72 0.36 4.09 5.52 1.00 3002 2088
Samples were drawn using sampling(NUTS). For each parameter, Bulk_ESS
and Tail_ESS are effective sample size measures, and Rhat is the potential
scale reduction factor on split chains (at convergence, Rhat = 1).Summary
summary(m3)
Family: gaussian
Links: mu = identity; sigma = identity
Formula: IQ ~ 0 + Group
Data: TwoGroupIQ (Number of observations: 89)
Samples: 4 chains, each with iter = 2000; warmup = 1000; thin = 1;
total post-warmup samples = 4000
Population-Level Effects:
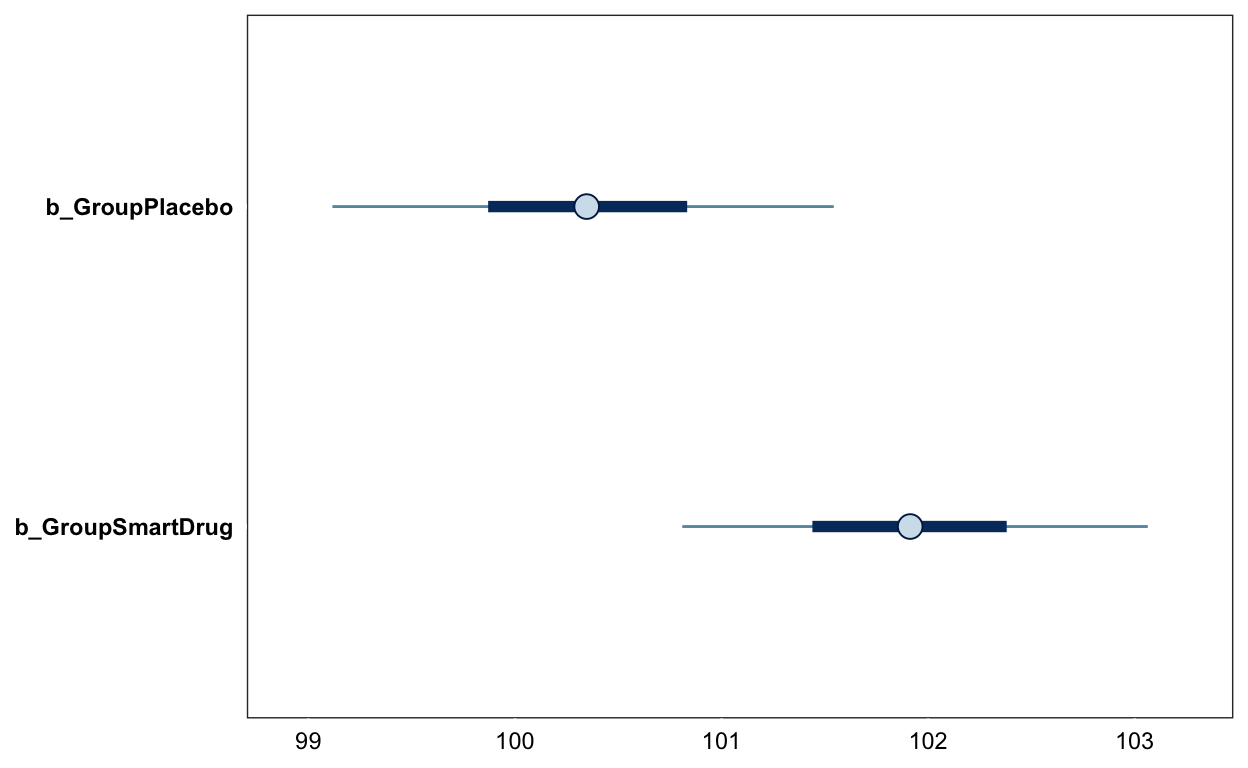
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS
GroupPlacebo 100.35 0.73 98.95 101.75 1.00 3914
GroupSmartDrug 101.92 0.69 100.62 103.28 1.00 4057
Tail_ESS
GroupPlacebo 3163
GroupSmartDrug 2954
Family Specific Parameters:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
sigma 4.71 0.36 4.07 5.45 1.00 3342 2697
Samples were drawn using sampling(NUTS). For each parameter, Bulk_ESS
and Tail_ESS are effective sample size measures, and Rhat is the potential
scale reduction factor on split chains (at convergence, Rhat = 1).mcmc_plot(m2, "b_GroupSmartDrug")

mcmc_plot(m3, "b")
Get Posterior Samples
samples_m3 <- posterior_samples(m3) %>%
transmute(Placebo = b_GroupPlacebo,
SmartDrug = b_GroupSmartDrug,
sigma = sigma)
samples_m3 <- samples_m3 %>%
mutate(diff = SmartDrug - Placebo,
effect_size = diff/sigma)
Get Posterior Samples
samples_m3 %>%
select(diff) %>%
median_qi()
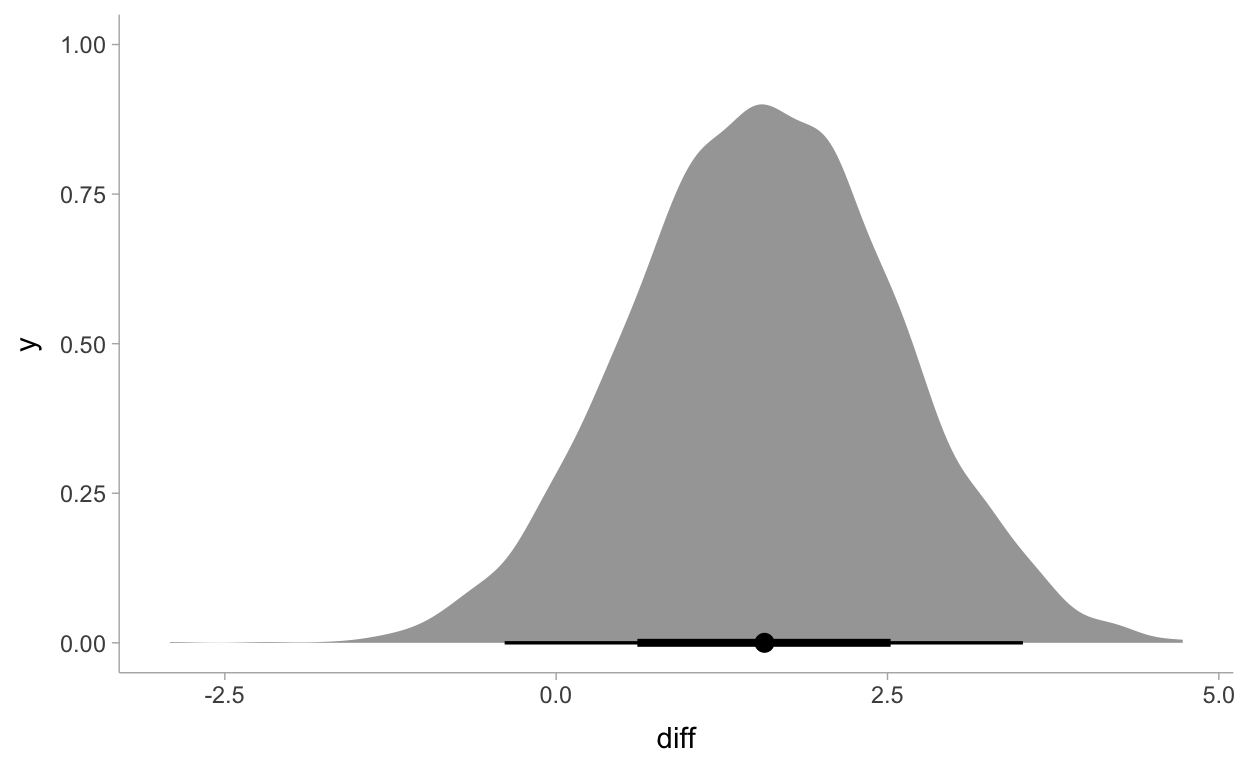
diff .lower .upper .width .point .interval
1 1.57189 -0.3887631 3.521777 0.95 median qisamples_m3 %>%
select(diff) %>%
ggplot(aes(x = diff)) +
stat_halfeye(point_interval = median_qi) +
theme_tidybayes()
samples_m3 %>%
select(effect_size) %>%
ggplot(aes(x = effect_size)) +
stat_halfeye(point_interval = median_qi) +
theme_tidybayes()

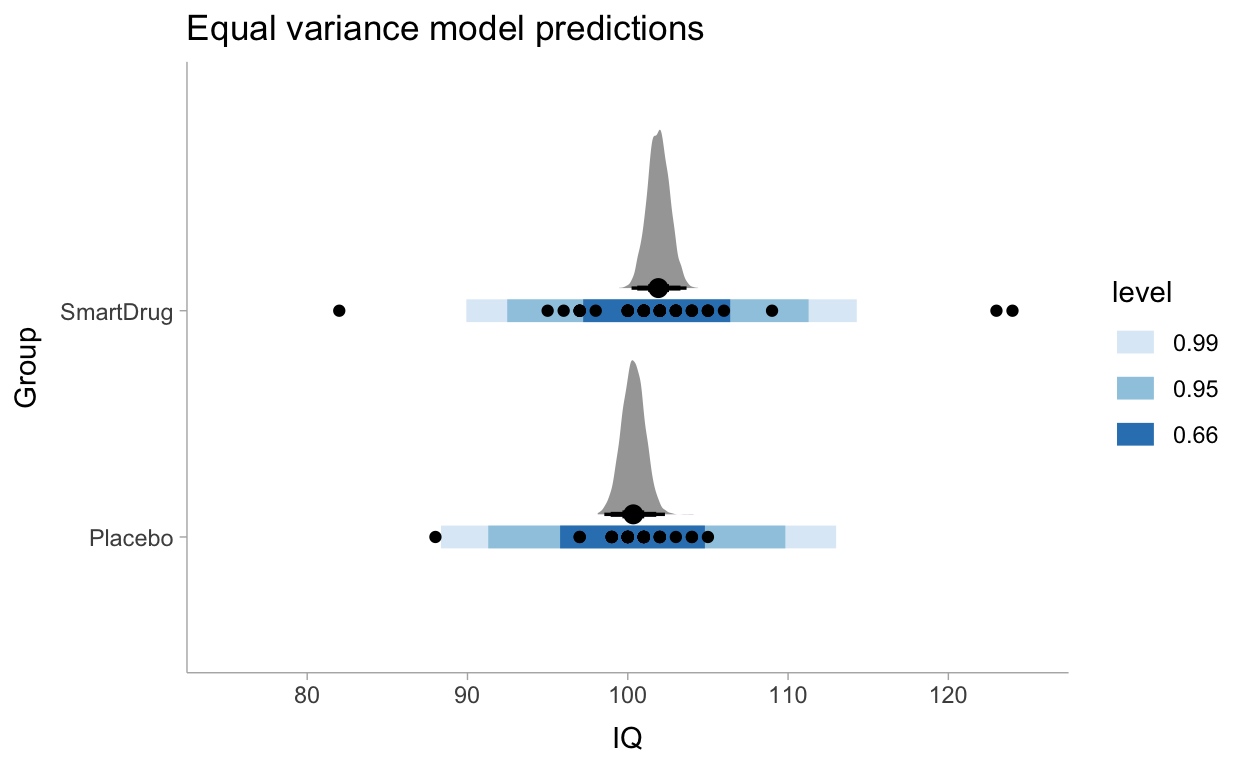
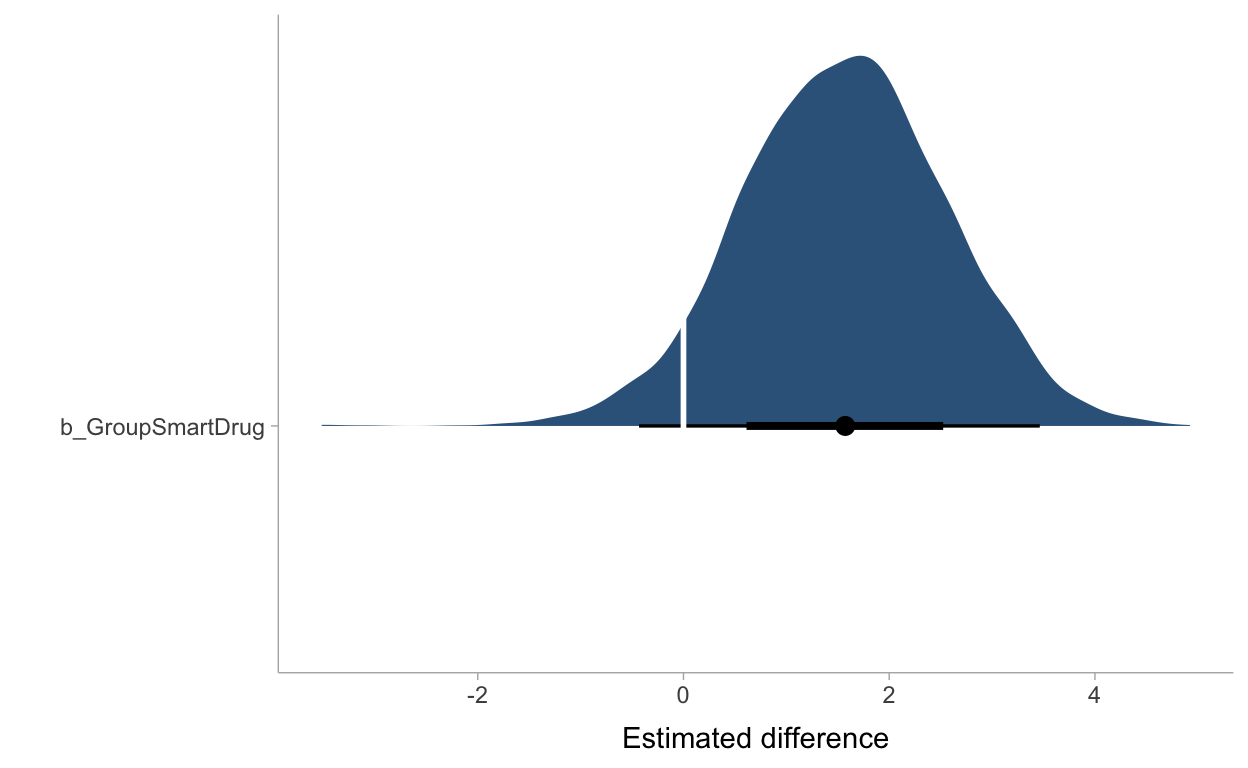
Diesmal ohne eigene Priors
fit_eqvar %>%
gather_draws(b_GroupSmartDrug) %>%
ggplot(aes(y = .variable, x = .value)) +
stat_halfeye(fill = "Steelblue4") +
geom_vline(xintercept = 0, color = "white", linetype = 1, size = 1) +
ylab("") +
xlab("Estimated difference") +
theme_tidybayes()
grid <- TwoGroupIQ %>%
modelr::data_grid(Group)
fits_IQ <- grid %>%
add_fitted_draws(fit_eqvar)
preds_IQ <- grid %>%
add_predicted_draws(fit_eqvar)
pp_eqvar <- TwoGroupIQ %>%
ggplot(aes(x = IQ, y = Group)) +
stat_halfeye(aes(x = .value),
scale = 0.7,
position = position_nudge(y = 0.1),
data = fits_IQ,
.width = c(.66, .95, 0.99)) +
stat_interval(aes(x = .prediction),
data = preds_IQ,
.width = c(.66, .95, 0.99)) +
scale_x_continuous(limits = c(75, 125)) +
geom_point(data = TwoGroupIQ) +
scale_color_brewer() +
labs(title = "Equal variance model predictions") +
theme_tidybayes()