Einführung
Bisher haben wir:
- Parameter geschätzt
- Binomial (Bernoulli) Modell
- Lineare Modelle mit kategorialem Prädiktor
- Posterior Verteilung(en) zusammengefasst
- Credible Intervals
- Highest Posterior Density Intervals
Wir würden gerne Aussagen über die Wahrscheinlichkeiten von Modellen machen:
- z.B. Modell 1 (M1) erklärt die Daten besser als Modell 2 (M2).
- M1 ist wahrscheinlicher als M2
Solche Aussagen sind Modellvergleiche.
Welche Methoden kennen Sie, um Modelle zu vergleichen?
- Diskutieren Sie anhand dieses Beispiels, wie Sie die Hypothese testen können, dass die Intervention einen Effekt hat.
library(tidyverse)
intervention <- rep(c('treat', 'control'), each = 5)
pre <- c(20, 10, 60, 20, 10, 50, 10, 40, 20, 10)
post <- c(70, 50, 90, 60, 50, 20, 10, 30, 50, 10)
dwide <- tibble(id = factor(1:10),
intervention, pre, post) %>%
mutate(diff = post - pre,
id = as_factor(id),
intervention = factor(intervention, levels = c("control", "treat")))
d <- dwide %>%
select(-diff) %>%
pivot_longer(cols = pre:post, names_to = "time", values_to = "score") %>%
mutate(time = as_factor(time))
Wir haben hier einen within (messwiederholten) Faktor (time), und einen between Faktor (intervention).
d %>%
ggplot(aes(time, score, color = intervention)) +
geom_line(aes(group = id), linetype = 1, size = 1) +
geom_point(size = 4) +
scale_color_viridis_d(end = 0.8)

Mögliche Methoden
- Hypothesentest, z.B.
t.test(diff ~ intervention,
data = dwide)
m1 <- lmer(score ~ intervention + time + (1|id),
data = d)
m2 <- lmer(score ~ intervention * time + (1|id),
data = d)
Modellvergleiche
Varianzaufklärung
Informationskriterien
Kreuzvalidierung
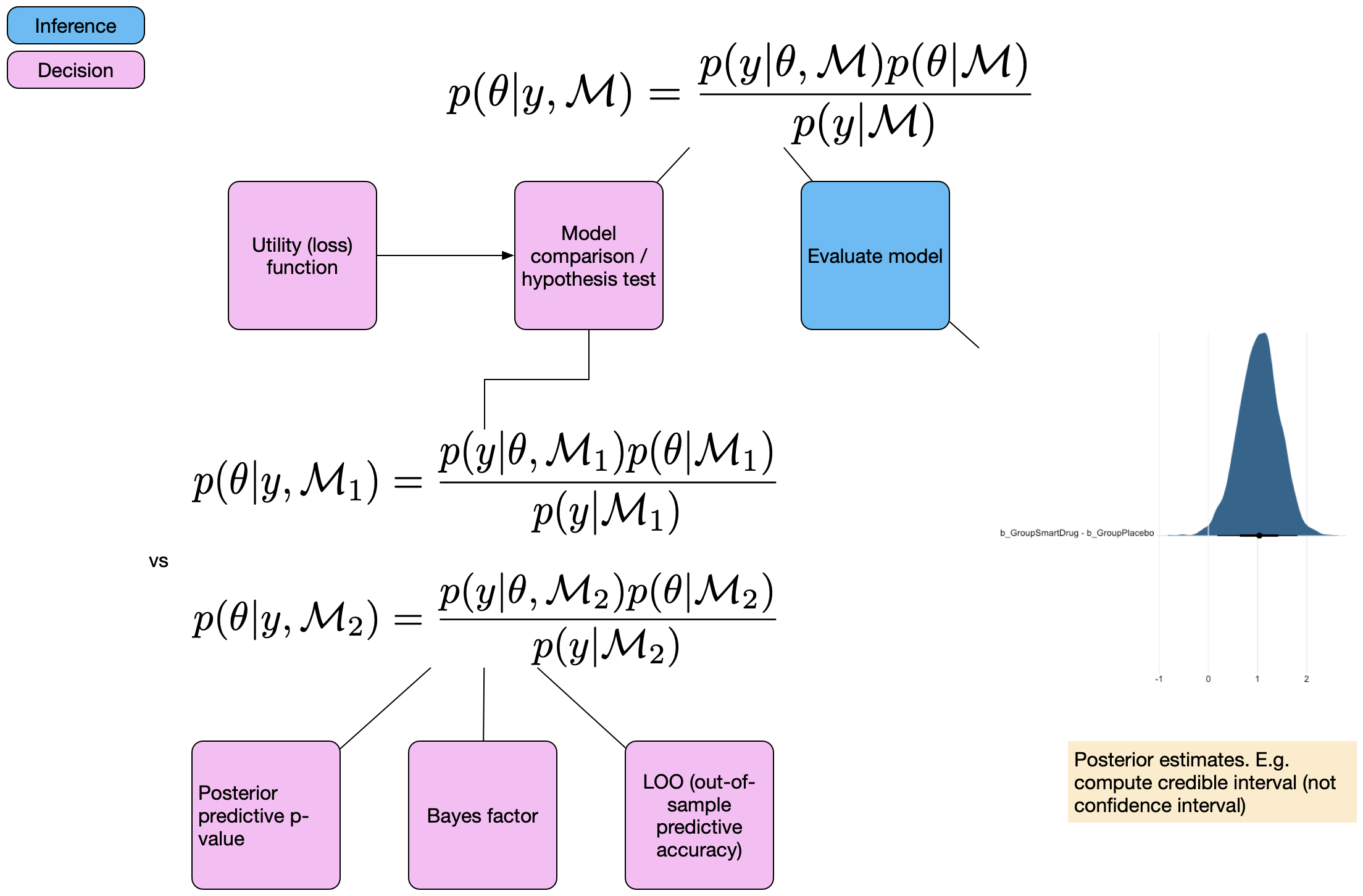
Wir lernen nun Methoden kennen, um mit Bayesianischen Modellen Modellvergleiche durchzuführen.
Bayesianische Modellvergleiche
Wir unterscheiden 3 verschiedene Methoden
Bayes Factors
Out of sample predictive accuracy:
Approximate leave-one-out cross validation (LOO)
Informationskriterien: widely application information criterion (WAIC)
- Posterior predictive checking: > Konstruktion einer Teststatistik und Vergleich der aktuellen Daten mit generierten Daten aus der Posterior Predictive Distribution.
Heute schauen wir uns die erste Methode (Bayes Factors) an. Diese sind unter Statistikern sehr umstritten. Psychologen wollen Hypothesen testen (Evidenz für/gegen Hypothesen), während in anderen Bereichen dies weniger verbreitet ist.


Bayes factor
Bayes’ Theorem, mit expliziter Abhängigkeit der Parameter \(\mathbf{\theta}\) vom Modells \(\mathcal{M}\):
\[ p(\theta | y, \mathcal{M}) = \frac{p(y|\theta, \mathcal{M}) p(\theta | \mathcal{M})}{p(y | \mathcal{M})}\]
\(\mathcal{M}\) bezieht sich auf ein bestimmtes Modell.
\(p(y | \mathcal{M})\) ist die Wahrscheinlichkeit der Daten (marginal likelihood), über alle möglichen Parameterwerte des Modells \(\mathcal{M}\) integriert. Wir haben sie anfangs als Normierungskonstante bezeichnet.
\[ P(\theta|Data) = \frac{ P(Data|\theta) * P(\theta) } {P(Data)} \]
\[ P(\theta|Data) \propto P(Data|\theta) * P(\theta) \]
\[ p(y | \mathcal{M}) = \int_{\theta}{p(y | \theta, \mathcal{M}) p(\theta|\mathcal{M})d\theta} \] Bei der Berechnung der Marginal Likelihood muss über alle unter dem Modell möglichen Werte von \(\theta\) gemittelt werden.
Komplexität
Marginal Likelihood wird auch Model Evidence genannt. Sie hängt davon ab, welche Vorhersagen ein Model machen kann (warum?).
Ein Modell, welches viele Vorhersagen machen kann, ist ein komplexes Modell. Die meisten Vorhersagen werden aber falsch sein (warum?).
Komplexität hängt unter anderem ab von:
Anzahl Parameter im Modell
Prior Verteilungen der Parameter
Bei frequentistischen Modellen spielt nur die Anzahl Parameter eine Rolle.
Prior Verteilungen
Uninformative Priors machen viele Vorhersagen, vor allem in Gegenden des Parameterraums, in denen die Lieklihood niedrig ist.
prior <- dbeta(x = p_grid, shape1 = 1, shape2 = 1)
compute_posterior(likelihood, prior)

prior <- dbeta(x = p_grid, shape1 = 20, shape2 = 20)
compute_posterior(likelihood, prior)

prior <- dbeta(x = p_grid, shape1 = 2, shape2 = 40)
compute_posterior(likelihood, prior)
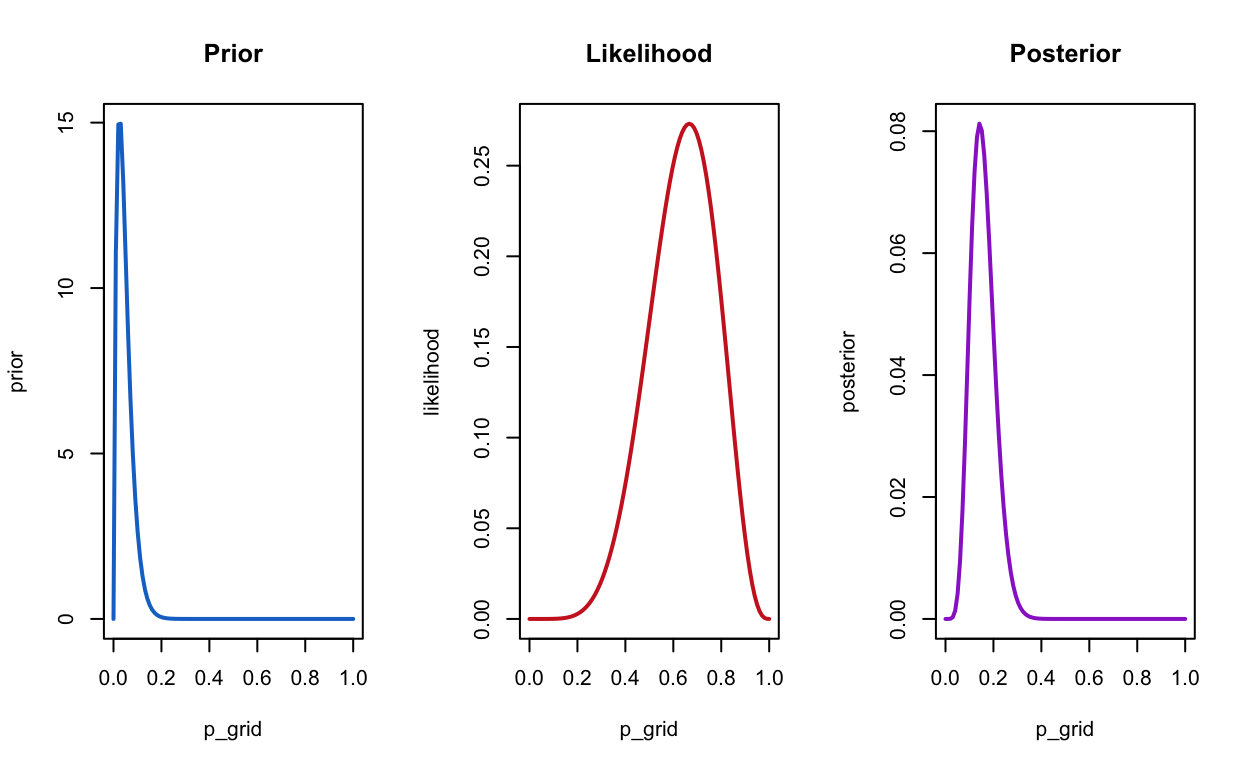
prior <- dbeta(x = p_grid, shape1 = 48, shape2 = 30)
compute_posterior(likelihood, prior)

Ockham’s Razor
Komplexe Modelle haben eine niedrigere Marginal Likelihood.
Wenn wir bei einem Vergleich mehrerer Modelle diejenigen Modelle mit höherer Marginal Likelihood bevorzugen, wenden wir das Prinzip der Sparsamkeit an.
Wir schreiben Bayes Theorem mit expliziten Modellen M1 und M2
\[ p(\mathcal{M}_1 | y) = \frac{P(y | \mathcal{M}_1) p(\mathcal{M}_1)}{p(y)} \]
und
\[ p(\mathcal{M}_2 | y) = \frac{P(y | \mathcal{M}_2) p(\mathcal{M}_2)}{p(y)} \]
Für Model \(\mathcal{M_m}\) ist die Posterior Wahrscheinlichkeit des Modells proportional zum Produkt der Marginal Likelihood und der A Priori Wahrscheinlichkeit.
Modellvergleich
Verhältnis der Modellwahrscheinlichkeiten:
\[ \frac{p(\mathcal{M}_1 | y) = \frac{P(y | \mathcal{M}_1) p(\mathcal{M}_1)}{p(y)}} {p(\mathcal{M}_2 | y) = \frac{P(y | \mathcal{M}_2) p(\mathcal{M}_2)}{p(y)}} \]
\(p(y)\) kann rausgekürzt werden.
\[\underbrace{\frac{p(\mathcal{M}_1 | y)} {p(\mathcal{M}_2 | y)}}_\text{Posterior odds} = \frac{P(y | \mathcal{M}_1)}{P(y | \mathcal{M}_2)} \cdot \underbrace{ \frac{p(\mathcal{M}_1)}{p(\mathcal{M}_2)}}_\text{Prior odds}\]
\(\frac{p(\mathcal{M}_1)}{p(\mathcal{M}_2)}\) sind die prior odds, und \(\frac{p(\mathcal{M}_1 | y)}{p(\mathcal{M}_2 | y)}\) sind die posterior odds.
Nehmen wir an, die Prior Odds seine \(1\), dann interessiert uns nur das Verhältnis der Marginal Likelihoods:
\[ \frac{P(y | \mathcal{M}_1)}{P(y | \mathcal{M}_2)} \]
Dieser Term ist der Bayes factor: der Term, mit dem die Prior Odds multipliziert werden. Er gibt an, unter welchem Modell die Daten wahrscheinlicher sind.
Wir schreiben \(BF_{12}\) - dies ist der Bayes factor für Modell 1 vs Modell 2.
\[ BF_{12} = \frac{P(y | \mathcal{M}_1)}{P(y | \mathcal{M}_2)}\]
\(BF_{12}\) gibt an, in welchem Ausmass die Daten \(\mathcal{M}_1\) bevorzugen, relativ zu \(\mathcal{M}_2\).
Beispiel: \(BF_{12} = 5\) heisst: die Daten sind unter Modell 1 5 Mal wahrscheinlicher als unter Modell 2
Klassifizierung
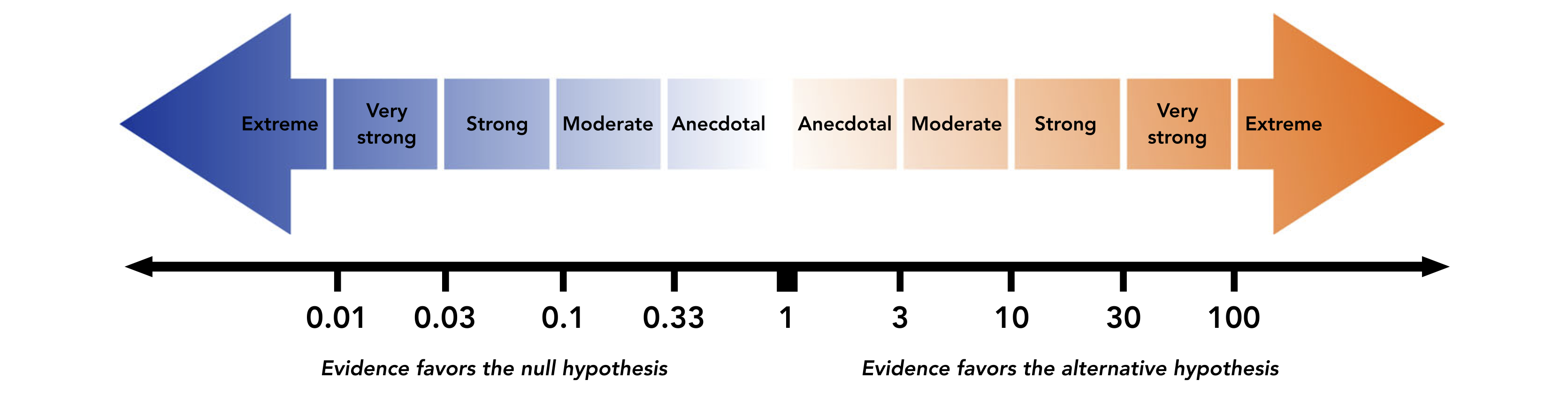
Bayes Factor
Sehr stark abhängig von den Prior Verteilungen der Parameter.
Ist nur für sehr simple Modelle einfach zu berechnen/schätzen.
Schätzmethoden
Savage-Dickey Density Ratio mit
Stan/brmsPackage BayesFactor (nur für allgemeine lineare Modelle)
JASP: IM Prinzip ein GUI für
BayesFactorBridge sampling mit
brms: schwieriger zu implementieren, aber für viele Modelle möglich.
Savage-Dickey Density Ratio
Wenn wir zwei genestete Modell vergleichen, wie z.B. ein Modell 1 mit einem frei geschätzten Parameter, und ein Nullmodell vergleichen, gibt es eine einfache Methode.
Unter dem Nullmodell (Nullhypothese): \(H_0: \theta = \theta_0\)
Unter Modell 1: \(H_0: \theta \neq \theta_0\)
Nun braucht der Parameter \(\theta\) unter Modell 1 eine Verteilung, z.B. \(\theta \sim \text{Beta}(1, 1)\)
Der Savage-Dickey Density Ratio Trick: wir betrachten nur Modell 1, und dividieren die Posterior Verteilung durch die Prior Verteilung an der Stelle \(\theta_0\).
Savage-Dickey Density Ratio
Wir schauen uns ein Beispiel aus Wagenmakers et al. (2010) an:
Sie beobachten, dass jemand 9 Fragen von 10 richtig beantwortet.
d <- tibble(s = 9, k = 10)
Unsere Frage: was ist die Wahrscheinlichkeit, dass das mit Glück ( \(\theta = 0.5\) ) passiert ist?


| x | Prior | Posterior | BF01 | BF10 |
|---|---|---|---|---|
| 0.5 | 1 | 0.107 | 0.107 | 9.309 |
Savage-Dickey Density Ratio
get_prior(m1_formula,
data = d)
prior class coef group resp dpar nlpar bound source
(flat) b default
(flat) b Intercept (vectorized)priors <- set_prior("beta(1, 1)",
class = "b",
lb = 0, ub = 1)
m1 <- brm(m1_formula,
prior = priors,
data = d,
sample_prior = TRUE, #<<
file = "models/05-m1")
summary(m1)
Family: binomial
Links: mu = identity
Formula: s | trials(k) ~ 0 + Intercept
Data: d (Number of observations: 1)
Samples: 4 chains, each with iter = 2000; warmup = 1000; thin = 1;
total post-warmup samples = 4000
Population-Level Effects:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
Intercept 0.83 0.10 0.58 0.97 1.00 1492 1345
Samples were drawn using sampling(NUTS). For each parameter, Bulk_ESS
and Tail_ESS are effective sample size measures, and Rhat is the potential
scale reduction factor on split chains (at convergence, Rhat = 1).Posterior summary
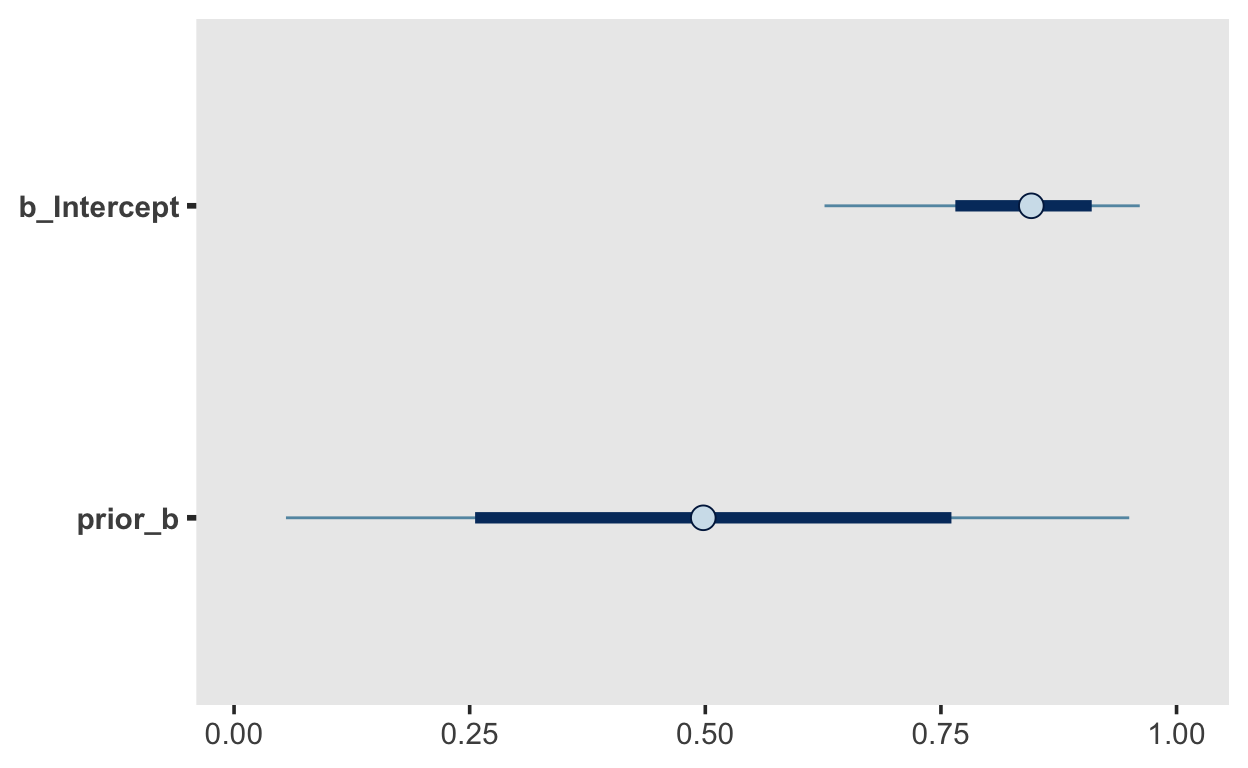
Prior und Posterior
.pull-left[samples <- m1 %>%
posterior_samples("b")
| b_Intercept | prior_b |
|---|---|
| 0.94 | 0.19 |
| 0.93 | 0.87 |
| 0.92 | 0.59 |
| 0.91 | 0.06 |
| 0.71 | 0.04 |
| 0.68 | 0.86 |
samples %>%
pivot_longer(everything(),
names_to = "Type",
values_to = "value") %>%
ggplot(aes(value, color = Type)) +
geom_density(size = 1.5) +
scale_color_viridis_d(end = 0.8) +
labs(x = bquote(theta), y = "Density") +
geom_vline(xintercept = .9)
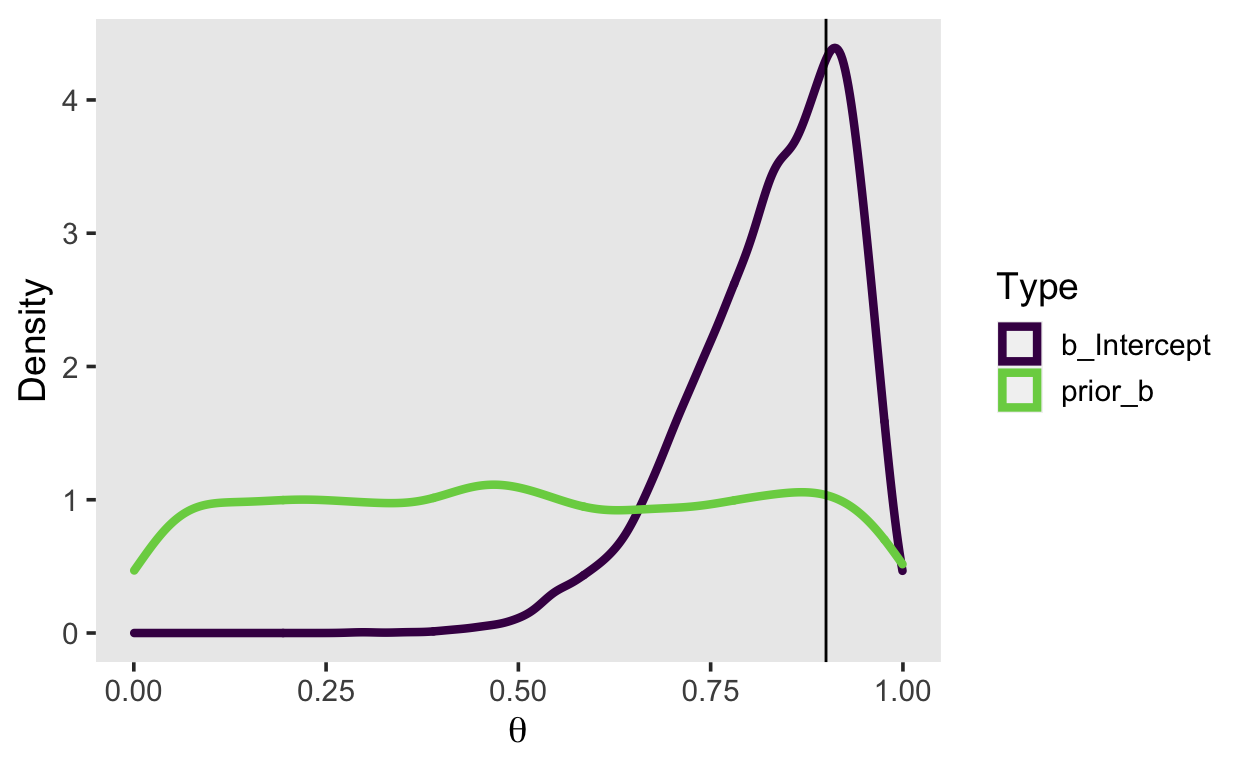
Savage-Dickey Density Ratio
Mit brms
h <- m1 %>%
hypothesis("Intercept = 0.5")
print(h, digits = 4)
Hypothesis Tests for class b:
Hypothesis Estimate Est.Error CI.Lower CI.Upper Evid.Ratio Post.Prob Star
1 (Intercept)-(0.5) = 0 0.3275 0.1048 0.0767 0.4721 0.1028 0.0932 *
---
'CI': 90%-CI for one-sided and 95%-CI for two-sided hypotheses.
'*': For one-sided hypotheses, the posterior probability exceeds 95%;
for two-sided hypotheses, the value tested against lies outside the 95%-CI.
Posterior probabilities of point hypotheses assume equal prior probabilities.Savage-Dickey Density Ratio
plot(h)
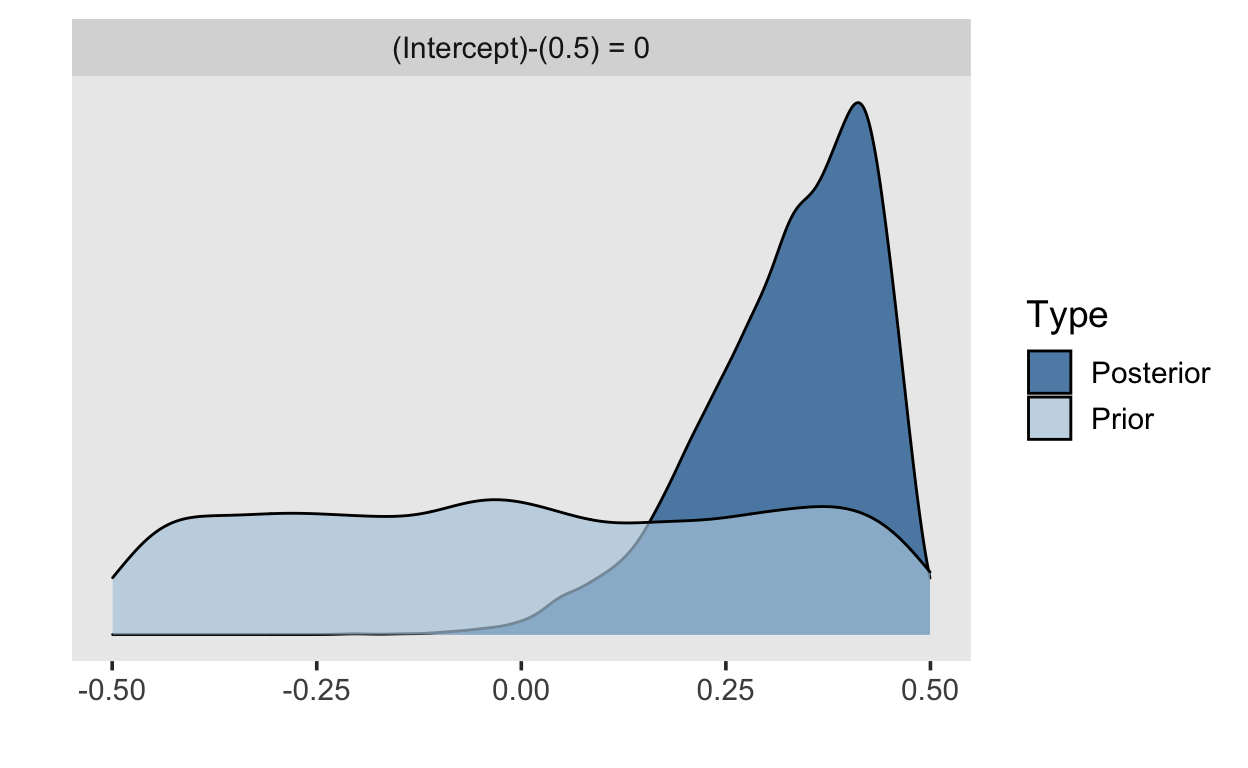
Bayes Factor für Vergleich von zwei Mittelwerten
smart = tibble(IQ = c(101,100,102,104,102,97,105,105,98,101,100,123,105,103,
100,95,102,106,109,102,82,102,100,102,102,101,102,102,
103,103,97,97,103,101,97,104,96,103,124,101,101,100,
101,101,104,100,101),
Group = "SmartDrug")
placebo = tibble(IQ = c(99,101,100,101,102,100,97,101,104,101,102,102,100,105,
88,101,100,104,100,100,100,101,102,103,97,101,101,100,101,
99,101,100,100,101,100,99,101,100,102,99,100,99),
Group = "Placebo")
TwoGroupIQ <- bind_rows(smart, placebo) %>%
mutate(Group = fct_relevel(as.factor(Group), "Placebo"))
t.test(IQ ~ Group,
data = TwoGroupIQ)
Welch Two Sample t-test
data: IQ by Group
t = -1.6222, df = 63.039, p-value = 0.1098
alternative hypothesis: true difference in means between group Placebo and group SmartDrug is not equal to 0
95 percent confidence interval:
-3.4766863 0.3611848
sample estimates:
mean in group Placebo mean in group SmartDrug
100.3571 101.9149 m2_formula <- bf(IQ ~ 1 + Group)
get_prior(m2_formula,
data = TwoGroupIQ)
prior class coef group resp dpar
(flat) b
(flat) b GroupSmartDrug
student_t(3, 101, 2.5) Intercept
student_t(3, 0, 2.5) sigma
nlpar bound source
default
(vectorized)
default
defaultm2 <- brm(m2_formula,
prior = priors,
data = TwoGroupIQ,
cores = parallel::detectCores(),
sample_prior = TRUE,
file = here::here("models/05-m2-iq-bf"))
summary(m2)
Family: gaussian
Links: mu = identity; sigma = identity
Formula: IQ ~ 1 + Group
Data: TwoGroupIQ (Number of observations: 89)
Samples: 4 chains, each with iter = 2000; warmup = 1000; thin = 1;
total post-warmup samples = 4000
Population-Level Effects:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS
Intercept 100.76 0.61 99.58 101.96 1.00 4374
GroupSmartDrug 0.76 0.71 -0.64 2.17 1.00 4267
Tail_ESS
Intercept 2977
GroupSmartDrug 3012
Family Specific Parameters:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
sigma 4.72 0.36 4.11 5.48 1.00 3654 2814
Samples were drawn using sampling(NUTS). For each parameter, Bulk_ESS
and Tail_ESS are effective sample size measures, and Rhat is the potential
scale reduction factor on split chains (at convergence, Rhat = 1).m2 %>%
mcmc_plot("GroupSmartDrug")
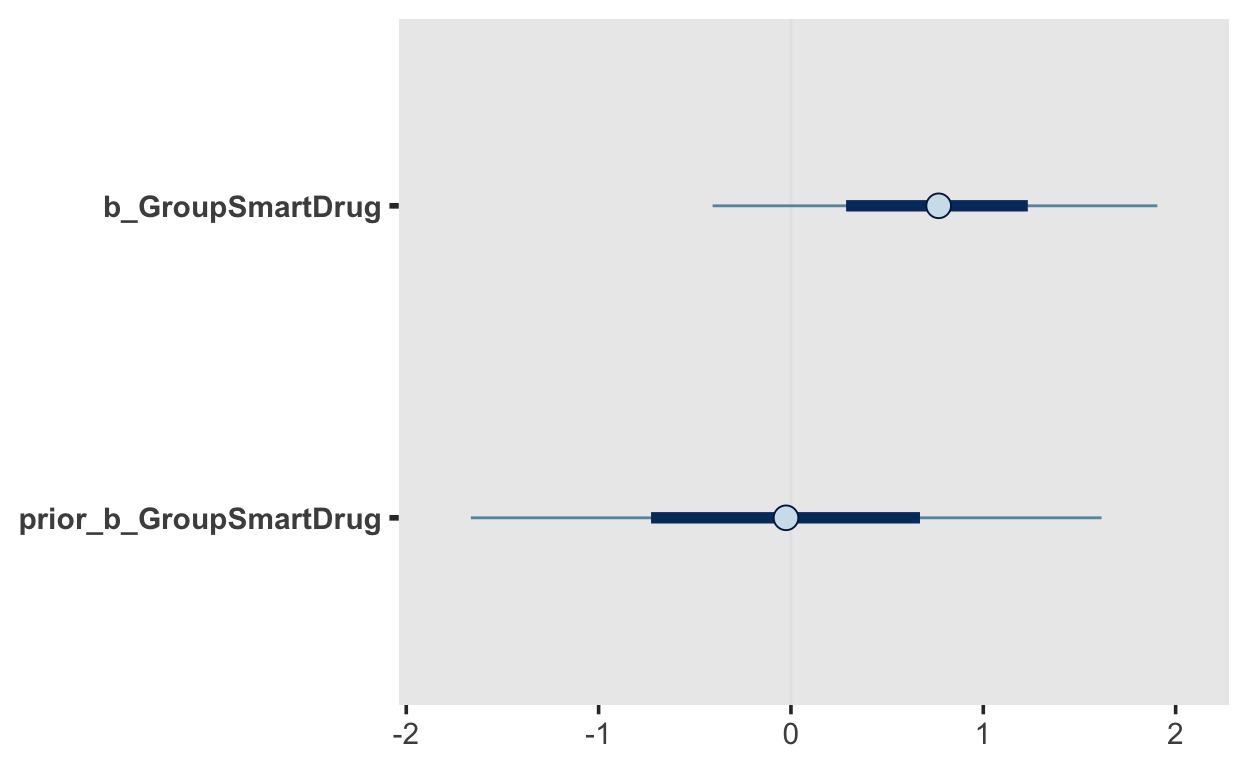
BF <- hypothesis(m2,
hypothesis = 'GroupSmartDrug = 0')
BF
Hypothesis Tests for class b:
Hypothesis Estimate Est.Error CI.Lower CI.Upper
1 (GroupSmartDrug) = 0 0.76 0.71 -0.64 2.17
Evid.Ratio Post.Prob Star
1 0.81 0.45
---
'CI': 90%-CI for one-sided and 95%-CI for two-sided hypotheses.
'*': For one-sided hypotheses, the posterior probability exceeds 95%;
for two-sided hypotheses, the value tested against lies outside the 95%-CI.
Posterior probabilities of point hypotheses assume equal prior probabilities.1/BF$hypothesis$Evid.Ratio
[1] 1.23144Speichern Sie den Dataframe mit der Funktion write_csv() TwoGroupIQ als CSV File.
write_csv(TwoGroupIQ, file = "SmartDrug.csv")
Laden Sie JASP herunter, und öffnen Sie das gespeicherte CSV File.
Führen Sie in JASP einen Bayesian t-Test durch.
- Warum erhalten Sie nicht denselben Bayes Factor wie JASP?
- Probieren Sie verschiedene Prior Verteilungen aus.
- Versuchen Sie, das Ergebnis in Worten zusammenzufassen. Was sagt der Bayes Factor aus?