Übung 4
Signal Detection Kennzahlen zusammenfassen.
In dieser Übung berechnen Sie aus den Daten von 15 Versuchspersonen aus dem PsychoPy Experiment die Signal Detection Kennzahlen \(d'\), \(k\) und \(c\). Anschliessen berechnen Sie Mittelwerte der drei Bedingungen für \(d'\) und \(c\) unter Berücksichtigung der Messwiederholung.
Die Aufgaben, die Sie bearbeiten sollen, finden Sie in einem gelben Kasten. Optionale Aufgaben sind in orangen Kästen.
In diesem File finden Sie Beispielscode. Manche Zeilen enthalten ___. Hier müssen Sie den Code vervollständigen.
Laden Sie bitte Ihre Lösung als R Skript bis Dienstag, 12.4.2022, um 00:30 Uhr, in den Order für Übung 4 auf ILIAS.
Nennen Sie Ihr File Matrikelnummer_Nachname_uebung-4.R.
Vorbereitung
Variablen bearbeiten
Zu factor konvertieren, etc.
Für jede Vpn in jeder der drei cue Bedingungen die verschiedenen Antworttypen zählen.
sdt_summary# A tibble: 170 × 4
# Groups: ID, cue [45]
ID cue type n
<fct> <fct> <chr> <int>
1 chch04 left CR 29
2 chch04 left FA 3
3 chch04 left Hit 7
4 chch04 left Miss 1
5 chch04 none CR 38
6 chch04 none FA 2
7 chch04 none Hit 34
8 chch04 none Miss 6
9 chch04 right CR 5
10 chch04 right FA 3
# … with 160 more rowsVon wide zu long konvertieren
sdt_summary <- sdt_summary |>
pivot_wider(names_from = type, values_from = n)sdt_summary# A tibble: 45 × 6
# Groups: ID, cue [45]
ID cue CR FA Hit Miss
<fct> <fct> <int> <int> <int> <int>
1 chch04 left 29 3 7 1
2 chch04 none 38 2 34 6
3 chch04 right 5 3 25 7
4 chmi14 left 21 10 5 3
5 chmi14 none 18 19 29 7
6 chmi14 right 3 4 26 4
7 J left 19 12 5 3
8 J none 23 16 33 6
9 J right 6 2 20 12
10 jh left 32 NA 5 3
# … with 35 more rowsFunktionen definieren
NAs ersetzen
Hit Rate und False Alarm Rate berechnen
sdt_summary <- sdt_summary |>
mutate(hit_rate = ___,
fa_rate = ___)Werte 0 und 1 korrigieren
Z-Transformation
sdt_summary# A tibble: 45 × 10
# Groups: ID, cue [45]
ID cue CR FA Hit Miss hit_rate fa_rate zhr zfa
<fct> <fct> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
1 chch04 left 29 3 7 1 0.875 0.0938 1.15 -1.32
2 chch04 none 38 2 34 6 0.85 0.05 1.04 -1.64
3 chch04 right 5 3 25 7 0.781 0.375 0.776 -0.319
4 chmi14 left 21 10 5 3 0.625 0.323 0.319 -0.460
5 chmi14 none 18 19 29 7 0.806 0.514 0.862 0.0339
6 chmi14 right 3 4 26 4 0.867 0.571 1.11 0.180
7 J left 19 12 5 3 0.625 0.387 0.319 -0.287
8 J none 23 16 33 6 0.846 0.410 1.02 -0.227
9 J right 6 2 20 12 0.625 0.25 0.319 -0.674
10 jh left 32 0 5 3 0.625 0.001 0.319 -3.09
# … with 35 more rowsSDT Kennzahlen berechnen
sdt_summary <- sdt_summary |>
mutate(dprime = ___,
k = ___,
c = ___) |>
mutate(across(c(dprime, k, c), round, 2))Variablen auswählen
sdt_final <- sdt_summary |>
select(ID, cue, dprime, k, c)Im finalen Datensatz haben wir nun d', k und c für jede Person in jeder Bedingung.
sdt_final# A tibble: 45 × 5
# Groups: ID, cue [45]
ID cue dprime k c
<fct> <fct> <dbl> <dbl> <dbl>
1 chch04 left 2.47 1.32 0.08
2 chch04 none 2.68 1.64 0.3
3 chch04 right 1.1 0.32 -0.23
4 chmi14 left 0.78 0.46 0.07
5 chmi14 none 0.83 -0.03 -0.45
6 chmi14 right 0.93 -0.18 -0.65
7 J left 0.61 0.29 -0.02
8 J none 1.25 0.23 -0.4
9 J right 0.99 0.67 0.18
10 jh left 3.41 3.09 1.39
# … with 35 more rowsWir erwarten, dass sich d' zwischen den Bedingungen nicht unterscheidet. k und c (bias) sollte sich hingegen zwischen den cue Bedingungen unterscheiden. Uns interessiert hier vor allem c: in der neutralen Bedingung sollte c etwa 0 sein, in der ‘left’ Bedingung sollte \(c > 0\) sein, und in der ‘right’ Bedingung sollte \(c < 0\) sein.
Versuchen Sie die untenstehende Grafiken für d' und c zu reproduzieren.
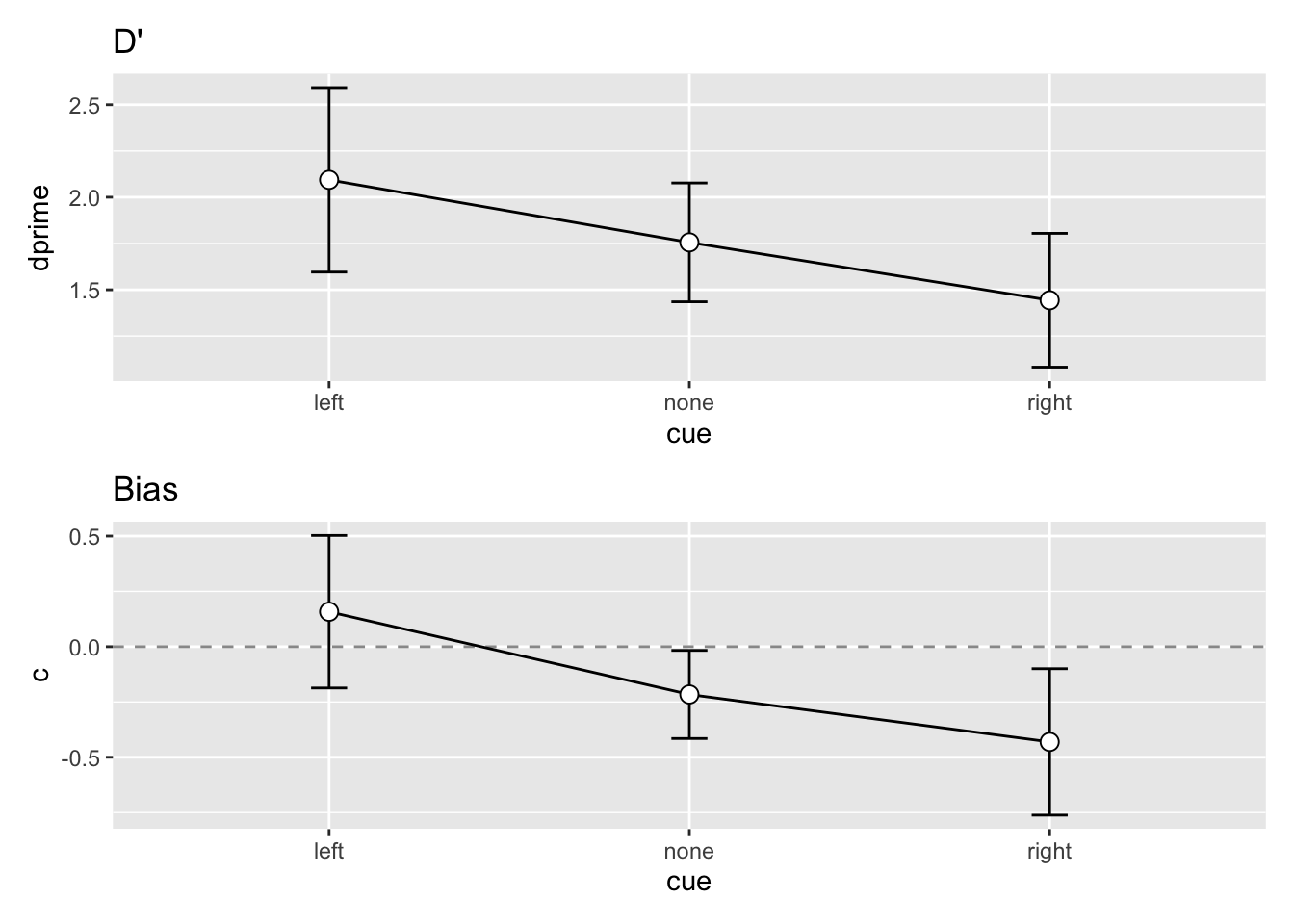
Sie brauchen zuerst eine (separate) Zusammenfassung der d' und c Werte, welche die Messwiederholung respektiert. Sie können dazu die Funktion summarySEwithin aus dem Rmisc Package verwenden.
Die Funktion braucht die Argumente measurevar, withinvars und idvar.
| Argument | Beschreibung |
|---|---|
measurevar |
Variable, für welche eine Messwiederholung vorliegt |
withinvars |
Messwiederholung |
idvar |
Identität der messwiederholten Einheit |
cs <- sdt_final |>
select(ID, cue, c) |>
___dprimes <- sdt_final |>
select(ID, cue, ___) |>
___Wenn Sie, wie ich, die Datensätze mit den Mittelwerten, Standardfehlern und \(95%\) Konfidenzintervallen primes und cs genannt haben, können Sie die Plots beispielsweise so erstellen.
cs |>
ggplot(aes(x = cue, y = c, group = 1)) +
geom_hline(yintercept = 0,
linetype = "dashed",
color = "grey60") +
geom_line() +
geom_errorbar(width = 0.1, aes(ymin = c - ci,
ymax = c + ci)) +
geom_point(shape = 21, size = 3, fill = "white") +
ggtitle("c (bias)")Falls Sie wollen, können Sie die individuellen c Schätzungen dem Plot hinzufügen, mit folgendem Code:
geom_jitter(aes(cue, c), data = sdt_final, width = 0.05)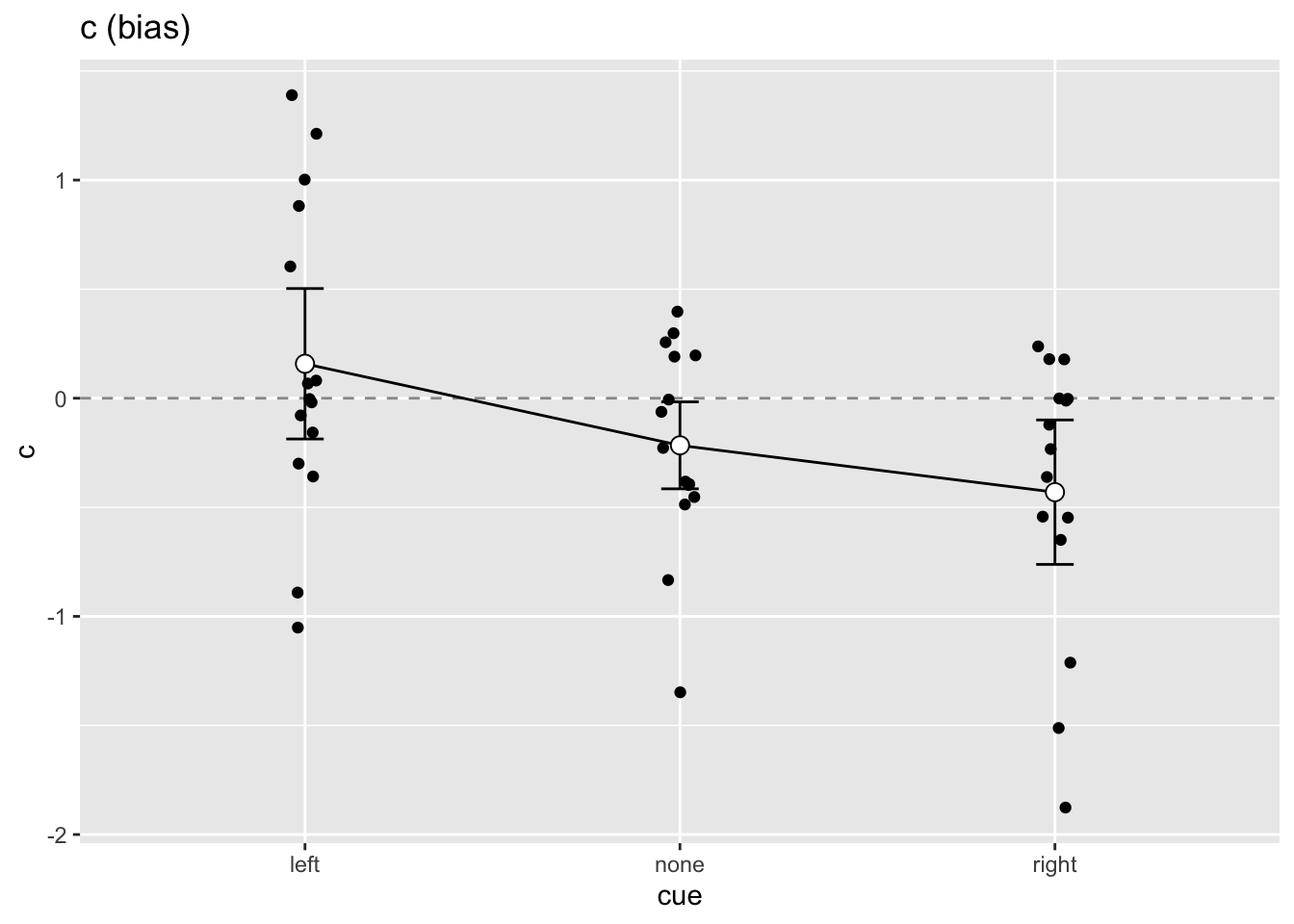
Reuse
Citation
@online{ellis2022,
author = {Andrew Ellis},
title = {Übung 4},
date = {2022-04-05},
url = {https://kogpsy.github.io/neuroscicomplabFS22//pages/exercises/exercise_04.html},
langid = {en}
}