10. Sitzung
Evidence accumulation models: II
Andrew Ellis
Neurowissenschaft Computerlab FS 22
2022-05-03
1
Simulating RT and choice data
Simulating RT and choice data
Load library
Parameters
Mit rdiffusion() können wir ein Experiment simulieren, bei dem die Fehler im Schnitt schneller als die korrekten Antworten sind, indem wir eine A Priori Präferenz für die Untergrenze definieren (z = 0.2).
Die 5 wichtigsten Argumente der Funktion sind:
n: Anzahl Zufallszahlen
a: boundary separation
v: drift rate
t0: non-decision time
z: biasSimulate
Rows: 500
Columns: 2
$ rt <dbl> 0.8207971, 0.5871117, 0.8568052, 0.5875147, 0.5460999, 0.5883…
$ response <fct> lower, upper, upper, upper, lower, lower, upper, upper, upper…Grafisch darstellen
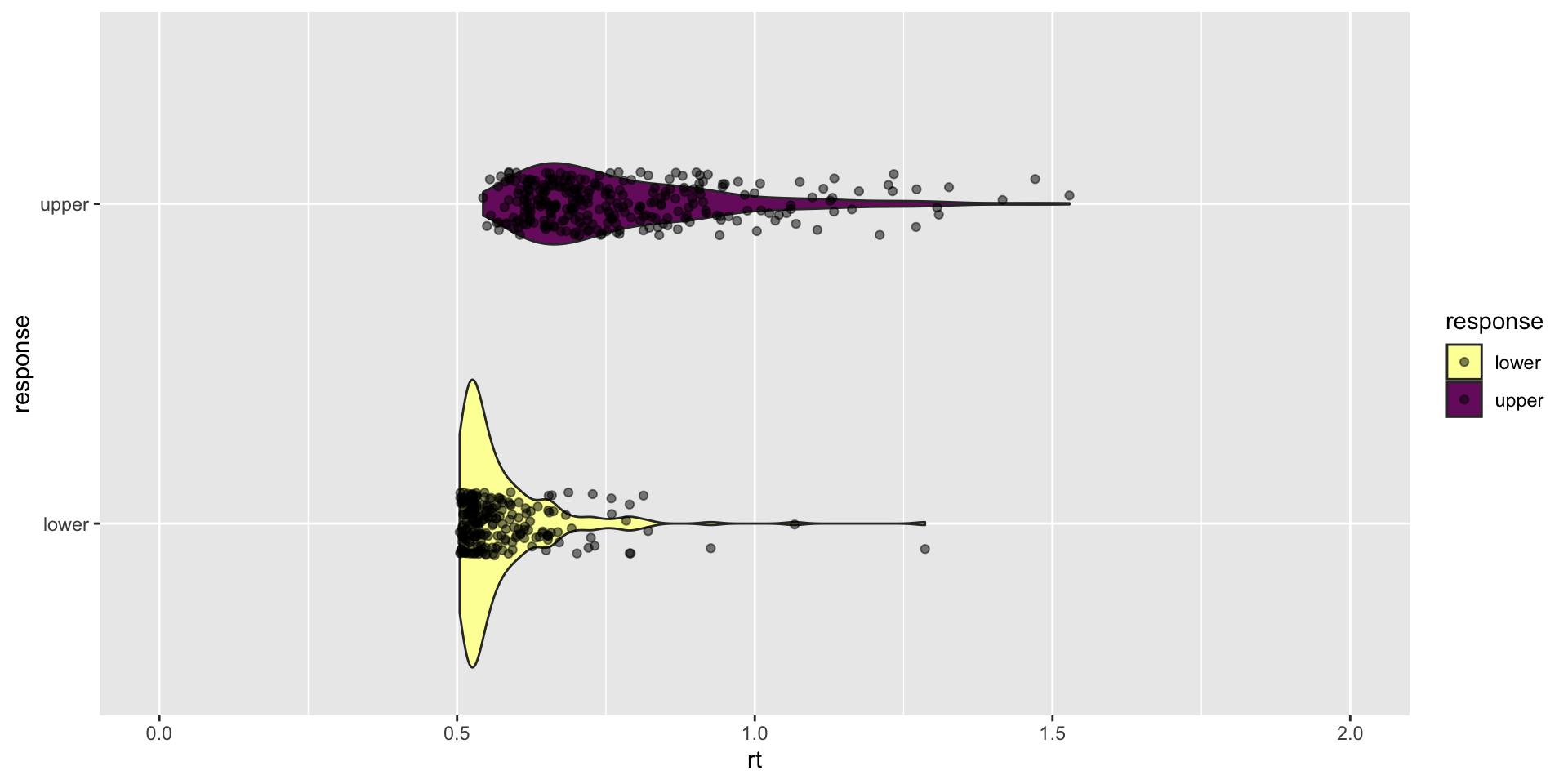
Effect of Bias
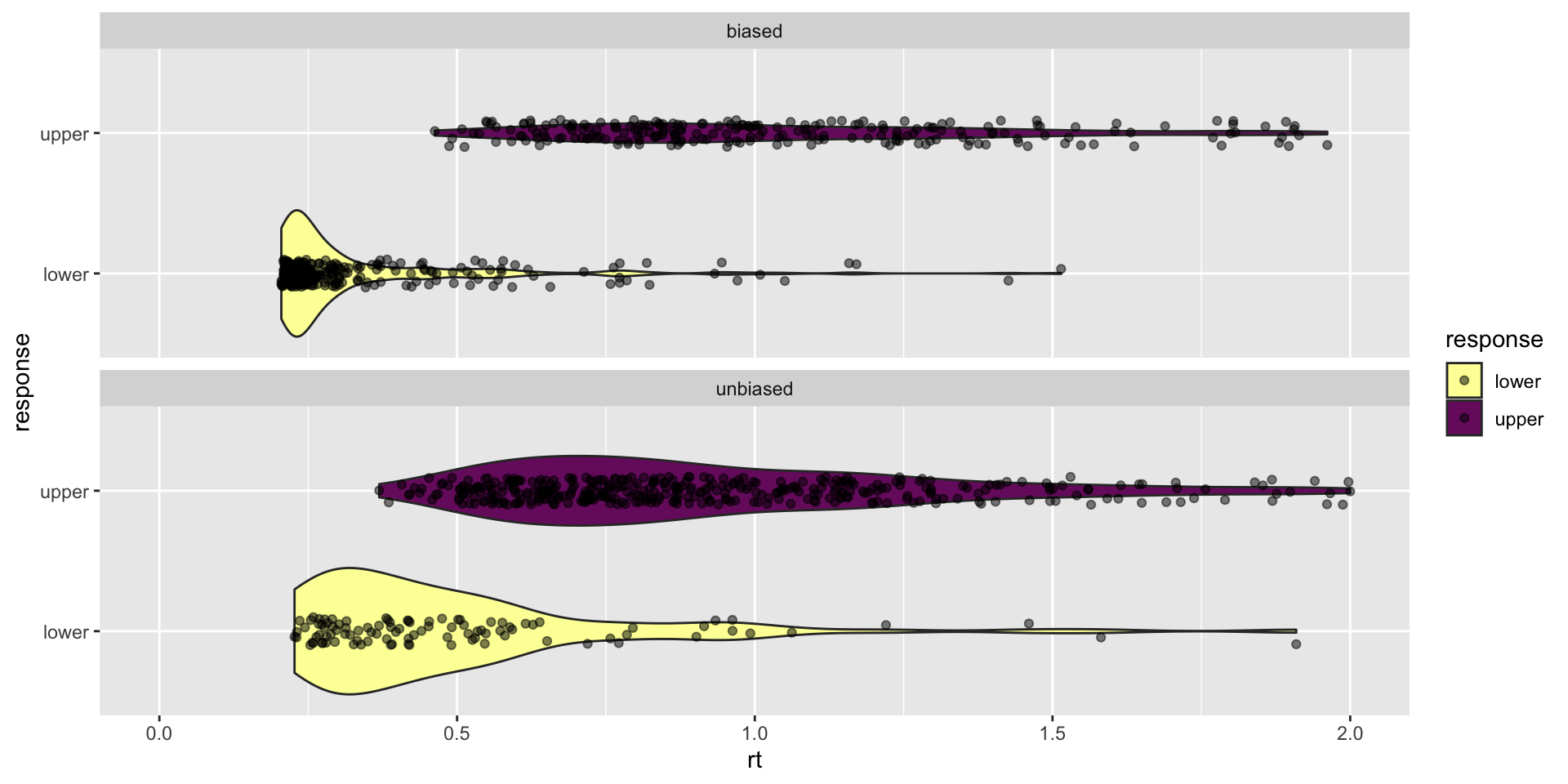
Zusammenfassen
2
Maximum likelihood estimation
Maximum Likelihood Schätzung
Die Likelihood - genauer gesagt die Likelihood-Funktion - ist eine Funktion, die angibt, wie wahrscheinlich es ist, einen bestimmten Satz von Beobachtungen mit einem bestimmten Modell zu erhalten.
Wir betrachten die Menge der Beobachtungen (Daten) als gegeben.
Nun überlegen wir, bei welchem Satz von Modellparametern wir die Daten am wahrscheinlichsten beobachten würden.
Wahrscheinlichkeit der Daten
Wenn wir eine Reihe von Datenpunkten haben (wie es in der Regel bei psychologischen Experimenten der Fall ist), können wir eine gemeinsame Wahrscheinlichkeit oder Wahrscheinlichkeitsdichte für die Daten in einem Datenvektor y erhalten, indem wir die einzelnen Wahrscheinlichkeiten oder Wahrscheinlichkeitsdichten miteinander multiplizieren, wobei wir davon ausgehen, dass die Beobachtungen in y unabhängig sind:
\[ f(\bf{y}|\bf{\theta} = \prod^k{f(y_k | \bf{\theta})}) \]
- \(k\) indiziert die Datenpunkte \(y_k\) im Vektor \(\bf{y}\).
Likelihood Funktion
Der Unterschied zwischen Wahrscheinlichkeit und Likelihood besteht darin, dass wir uns hier für die verschiedenen mögliche Parameterwerte beziehen, und die Likelihood-Funktion sagt uns, wie wahrscheinlich jeder dieser Parameterwerte angesichts der von uns beobachteten Daten ist.
\[ L(\bf{\theta} | \bf{y}) = \prod^k{L(\bf{\theta} | y_k}) \]
Beisiel: Binomial likelihood
Wir werfen eine Münze:
- zwei mögliche Ergebnisse (Kopf und Zahl),
- feste Anzahl von “Versuchen” (100 Münzwürfe)
- feste Wahrscheinlichkeit für “Erfolg” (d. h. Kopf)
Wahrschienlichkeit der Daten gegeben \(p = 0.6\)
Beispiel Kartenspiel zwischen 2 Spielern
- Spieler A gewinnt 6 Mal in 9 Spielen:
- Wir definieren einen Vektor mit 100 möglichen Parameterwerten:
- Nun berechnen wir die Wahrscheinlichkeit der Daten, gegeben die Parameterwerte:
Beispiel Kartenspiel zwischen 2 Spielern
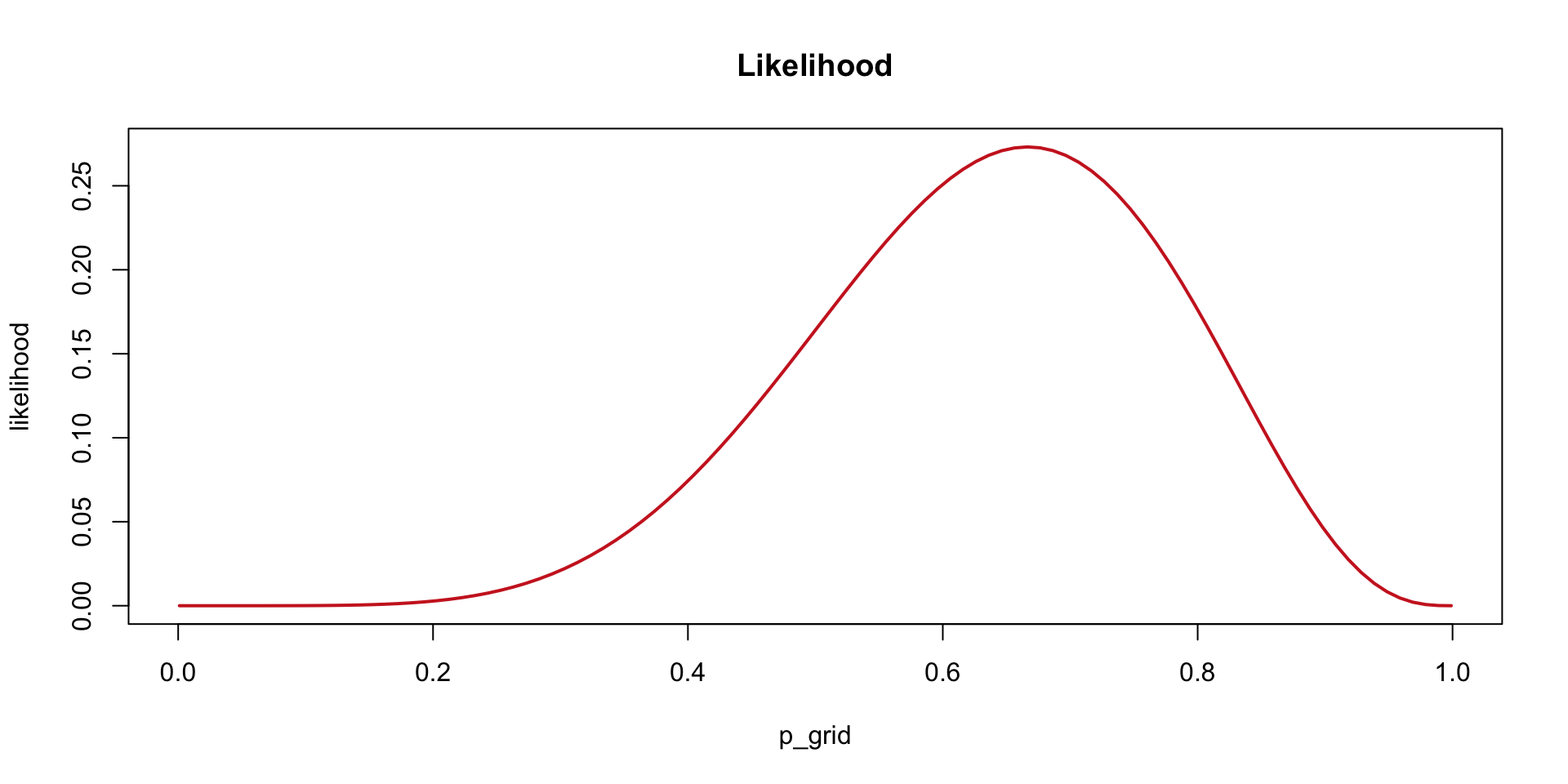
- Index des Parameterwertes, welcher die Likelihood maximiert:
- Parameterwert andiesem Index:
MLE: Negative Log Likelihood minimieren
- Definiere eine Funktion, welche die negative log Wahrscheinlichkeitsfunktion für einen bestimmten Wert von p berechnet.
- Suche nach dem Wert von p, für den diese Funktion minimiert wird.
Binomial Likelihood
\[ f(k | p_{heads}, N) = \binom{N}{k} p_{heads}^k (1-p_{heads}^{N-k}) \] \[ lnL(\bf{\theta} | \bf{y}) = \sum^k_{k=1}{ln L(\bf{\theta} | y_k}) \]
Funktion definieren
Minimieren
$minimum
[1] 2.530081
$estimate
[1] 0.4799995
$gradient
[1] 0
$code
[1] 1
$iterations
[1] 8