Good Practices in der Datenverarbeitung
Herausforderungen in der Arbeit mit neurowissenschaftlichen Daten
In der neurowissenschaftlichen Forschung werden zunehmend sehr grosse und komplexe Datensätze generiert. Daten aus unterschiedlichen Datenerhebungsverfahren sollen miteinander verknüpft (aggregiert) werden, um neue Erkenntnisse zu gewinnen. Eine sehr häufige Kombination sind beispielsweise Verhaltens- und Bildgebungsdaten, wie es in vielen fMRI-Studien der Fall ist. Das erfordert Kenntnisse der unterschiedlichen Formate und Eigenschaften dieser Daten sowie Programmierkenntnisse um diese Daten möglichst automatisiert vorzuverarbeiten, zu verknüpfen, visualisieren und analysieren.
Lesen Sie den untenstehenden Abschnitt aus Pierré et al. (2024). Besprechen Sie in Gruppen, welche spezifischen Herausforderungen Datenmanagement, -vorverarbeitung und -analyse in den Neurowissenschaften bestehen.
Increasing complexity of neuroscience data
Over the past 20 years, neuroscience research has been radically changed by two major trends in data production and analysis.
First, neuroscience research now routinely generates large datasets of high complexity. Examples include recordings of activity across large populations of neurons, often with high resolution behavioral tracking (Steinmetz et al., 2019; Stringer et al., 2019; Mathis et al., 2018; Siegle et al., 2021; Koch et al., 2022), analyses of neural connectivity at high spatial resolution and across large brain areas (Scheffer et al., 2020; Loomba et al., 2022), and detailed molecular profiling of neural cells (Yao et al., 2023; Langlieb et al., 2023; Braun et al., 2022; Callaway et al., 2021). Such large, multi-modal data sets are essential for solving major questions about brain function (Brose, 2016; Jorgenson et al., 2015; Koch and Jones, 2016).
Second, the collection and analysis of such datasets requires interdisciplinary teams, incorporating expertise in systems neuroscience, engineering, molecular biology, data science, and theory. These two trends are reflected in the increasing numbers of authors on scientific publications (Wareham, 2016), and the creation of mechanisms to support team science by the NIH and similar research funding bodies (Cooke and Hilton, 2015; Volkow, 2022; Brose, 2016).
There is also an increasing scope of research questions that can be addressed by aggregating “open data” from multiple studies across independent labs. Funding agencies and publishers have begun to aggressively promote data sharing and open data, with the goals of improving reproducibility and increasing data reuse (Dallmeier-Tiessen et al., 2014; Tenopir et al., 2015; Pasquetto et al., 2017). However, open data may be unusable if scattered in a wide variety of naming conventions and file formats lacking machine-readable metadata.
Big data and team science necessitate new strategies for how to best organize data, with a key technical challenge being the development of standardized file formats for storing, sharing, and querying datasets. Prominent examples include the Brain Imaging Data Structure (BIDS) for neuroimaging, and Neurodata Without Borders (NWB) for neurophysiology data (Teeters et al., 2015; Gorgolewski et al., 2016; Rübel et al., 2022; Holdgraf et al., 2019). The Open Neurophysiology Environment (ONE), best known from adoption by The International Brain Laboratory (The International Brain Laboratory et al., 2020, 2023), has a similar application domain to NWB, but a highly different technical design. (…)
These initiatives provide technical tools for storing and accessing data in known formats, but more importantly provide conceptual frameworks with which to standardize data organization and description in an (ideally) universal, interoperable, and machine-readable way. Pierré et al. (2024) (Preprint)
Datenquellen, Datenformate und Interdisziplinariät
Neurowissenschaftliche Daten von Verhaltensdaten zu Bildgebungsdaten stammen aus unterschiedlichsten Quellen und haben alle spezifische Eigenschaften, die in der Datenverarbeitung berücksichtigt werden müssen. Das bedeutet, dass sehr unterschiedliche Formate miteinander verknüpft werden müssen für die Analyse. Bei der Generierung von Daten sind oft auch Personen aus unterschiedlichen Fachrichtungen beteiligt, welche andere Hintergründe bezüglich Datenmanagement haben (z.B. bei fMRI Experimenten Fachpersonen aus den Bereichen Radiologie, Physik, Neurowissenschaften, Psychologie, Medizin, etc.). Auch bei der Analyse sind oft verschiedene Personen beteiligt, die alle darauf angewiesen sind, den Datensatz zu verstehen.
Grosse und komplexe Datensätze
Neurowissenschaftliche Datensätze sind oft sehr gross (pro Versuchsperson oft mehrere Gigabytes) und für die Speicherung und das Safety Back-up wird also viel Speicherplatz benötigt (mehrere Terrabytes). Komplexe Datensätze bedeutet, dass eine gute Dokumentation nötig ist, welche Variable welche Bedeutung hat und wo gegebenenfalls Änderungen gemacht wurden.
Open Science
Falls dabei keine Persönlichkeitsrechte verletzt werden, sollten Daten möglichst zugänglich gemacht werden, um einerseits transparente Resultate zu ermöglichen und andererseits kann so ein Datensatz auch von anderen Forschenden für weitere Fragestellung verwendet werden. Dies ist aber nur möglich, wenn der Datensatz für Aussenstehende verständlich abgespeichert wurde.
Zahlreiche Zwischenschritte
Von der Datenerhebung bis zur Datenanalyse werden die Daten oft ausführlich vorverarbeitet. Dazu gehören u.a. folgende Schritte (wobei jeder eigene Fehlerquellen beinhalten kann):
- Importieren der Rohdaten
- Verändern des Formats (z.B.
.dcm->.niiin fMRI) - Identifikation von Missings
- Ausschluss von ungültigen Messungen
- Anonymisierung
- Weglassen nicht benötigter Datenpunkte
- Fehlende Werte ergänzen
- Duplizierungen identifizieren und löschen
- Metadaten hinzufügen
- Datensätze zusammenfügen
- Spalten teilen
- Variablen umbenennen
- Variablen normalisieren/standardisieren
- Variablen verändern (z.B. ms zu sec)
- Variablentypen anpassen
- Variablen recodieren
- Neue Variablen erstellen
- Neuer Datensatz abspeichern
Bevor mit einem Datensatz gearbeitet wird, empfiehlt es sich den Datensatz anzuschauen und folgendes zu identifizieren:
- In welchem Dateiformat ist der Datensatz gespeichert? z.B. in
.csv,.xlsxoder anderen?` - In welchem Datenformat ist der Datensatz geordnet? (
longoderwideodermixed?) - Gibt es ein
data dictionnarymit Erklärungen zu den Variablen?
Verschiedene Standards
Es gibt zahlreiche Bemühungen Datenmanagement in den Neurowissenschaften zu vereinheitlichen. Viele verfügbare Standards und Tools machen die Datenverarbeitung aber nicht nur einfacher. Im nächsten Kapitel finden Sie deshalb eine Übersicht von Ansätzen, die uns sinnvoll und allgemein anwendbar erscheinen und sich in unseren Labors bewährt haben.
Good Practices in der Datenverarbeitung
Im Folgenden wird auf einige wichtige Good Practices in der Datenverarbeitung eingegangen. Wichtig sind hier die Reproduzierbarkeit der Datenverarbeitungs- und Datenanalysepipelines sowie die Sicherstellung der Datenqualität.
Reproduzierbarkeit
Die Replikationskrise hat in der Psychologie, aber auch in den kognitiven Neurowissenschaften ein Umdenken ausgelöst. Mit dem Ziel nachhaltigere Forschungsergebnisse zu erreichen sind verschiedene Begriffe wie Reproduzierbarkeit und Replizierbarkeit zu wichtigen Schlagworten geworden. Die Begrifflichkeiten werden verwirrenderweise aber oft unterschiedlich definiert und verwendet (Plesser (2018)).
Replizierbarkeit (replicability) bedeutet, dass ein Experiment von einer anderen Forschungsgruppe mit einer neuen Stichprobe durchgeführt werden kann, und ähnliche oder dieselben Resultate hervorbringt, wie die Originalstudie. Wird eine Studie mehrmals repliziert, steigt die Wahrscheinlichkeit, dass kein Zufallsbefund vorliegt.
Replicability refers to the ability of a researcher to duplicate the results of a prior study if the same procedures are followed but new data are collected. Cacioppo et al. (2015)
Reproduzierbarkeit (reproducibility) hängt eng mit der Replizierbarkeit zusammen. Der Begriff wird teilweise sehr allgemein verwendet, und bedeutet so dass Forschungsergebnisse wiederholt gefunden werden. Reproduzierbarkeit im engeren Sinn hingegen bezieht sich darauf, ob die durchgeführte Analyse wiederholt werden kann. Die Reproduzierbarkeit ist somit hoch, wenn Forschende die Daten und Datenanalyseskripts bereitstellen und andere Forschende damit dieselben Analysen durchführen können und zu gleichen Resultaten kommen.
Reproducibility refers to the ability of a researcher to duplicate the results of a prior study using the same materials as were used by the original investigator. That is, a second researcher might use the same raw data to build the same analysis files and implement the same statistical analysis in an attempt to yield the same results…. Reproducibility is a minimum necessary condition for a finding to be believable and informative. Cacioppo et al. (2015)
Um die Begriffe zusammenzufassen schlugen Goodman, Fanelli, and Ioannidis (2016) vor von
- Reproduzierbarkeit der Methoden (Daten und Prozesse können exakt wiederholt werden)
- Reproduzierbarkeit der Resultate (andere Studien kommen auf dieselben Resultate) und
- Reproduzierbarkeit der wissenschaftlichen Schlussfolgerung (bei Repetition der Analyse oder der Experimente werden dieselben Schlüsse gezogen)
zu sprechen. Die Reproduzierbarkeit von Resultaten und Schlussfolgerungen ist hier nicht klar abgrenzbar vom Begriff der Replizierbarkeit. Grundsätzlich besteht das Ziel, dass in der Forschung möglichst viel Evidenz für eine Schlussfolgerung gesammelt werden kann. Dies gelingt, wenn die Prozesse transparent, fehlerfrei und wiederholbar sind.
Hindernisse bei der Reproduzierbarkeit
In diesem Kurs beschäftigen wir uns vor allem mit der Reproduzierbarkeit der Methoden als Grundlage für solide Erkenntnisgewinne. Reproduzierbarkeit kann laut Nosek et al. (2022) vor allem aus zwei Gründen nicht gegeben sein: Weil die Daten/Skripte nicht zur Verfügung stehen, oder weil diese Fehler enthalten:
In principle, all reported evidence should be reproducible. If someone applies the same analysis to the same data, the same result should occur. Reproducibility tests can fail for two reasons. A process reproducibility failure occurs when the original analysis cannot be repeated because of the unavailability of data, code, information needed to recreate the code, or necessary software or tools. An outcome reproducibility failure occurs when the reanalysis obtains a different result than the one reported originally. This can occur because of an error in either the original or the reproduction study. Nosek et al. (2022)
Führt die Reproduktion nicht zum selben Resultat, löst das Zweifel am Forschungsergebnis aus. Wenn die Reproduzierbarkeit am Prozess scheitert, etwa weil die Daten nicht vorhanden sind, kann kein Schluss gezogen werden, ob die Resultate stimmen.
Achieving reproducibility is a basic foundation of credibility, and yet many efforts to test reproducibility reveal success rates below 100%. … Whereas an outcome reproducibility failure suggests that the original result may be wrong, a process reproducibility failure merely indicates that the original result cannot be verified. Either reason challenges credibility and increases uncertainty about the value of investing additional resources to replicate or extend the findings (Nuijten et al. 2018). Sharing data and code reduces process reproducibility failures (Kidwell et al. 2016), which can reveal more outcome reproducibility failures (Hardwicke et al. 2018, 2021; Wicherts et al. 2011). Nosek et al. (2022)
Das Teilen von Daten und Datenverarbeitungsskripten erhöht demnach die Wahrscheinlichkeit, dass mögliche Fehler im Prozess gefunden werden, da auch andere Forschende die Daten/Skripts verwenden können. Das ist vorerst unangenehm, gehört aber zum wissenschaftlichen Prozess dazu. Die Normalisierung einer Fehlerkultur in diesem Bereich würde indirekt auch die Replizierbarkeit von Ergebnissen erhöhen.
Neuroimaging experiments result in complicated data that can be arranged in many different ways. So far there is no consensus how to organize and share data obtained in neuroimaging experiments. Even two researchers working in the same lab can opt to arrange their data in a different way. Lack of consensus (or a standard) leads to misunderstandings and time wasted on rearranging data or rewriting scripts expecting certain structure. BIDS Website (2024)
Tools für Reproduzierbarkeit
Für reproduzierbare Forschung gibt es inzwischen viele gute Tools:
OSF: Website der Open Science Foundation
Eine kostenfreie und unkomplizierte Möglichkeit Daten und Skripts zu teilen, und diese in Projekten abzulegen. Es lässt sich dafür sogar ein doi erstellen. Auch Formulare für eine Präregistration sind hier zu finden.
FAIR Guiding Principles
Beim Veröffentlichen von wissenschaftlichen Artikeln ist es empfohlen, die Daten (falls Anonymisierung möglich) sowie die Analyse-Skripte mitzuveröffentlichen.
Für Datensätze gelten die FAIR Guiding Principles (Wilkinson et al. (2016)):
F indability: Es ist klar unter welchen Umständen und wie die Daten zugänglich sind
A ccessibility: Daten sind zugänglich bzw. es ist klar wo sie zu finden wären
I nteroperability: Verwendbare Datenformate/strukturen
R eusability: gute Beschreibung des Datensatzes/der enthaltenen Variablen
BIDS: Brain Imaging Data Structure
Für Neuroimaging-Daten gibt es beispielsweise vorgegebene Konventionen, wie ein Datensatz und die Verarbeitungsskripts abgespeichert werden. Ein Beispiel dafür ist Brain Imaging Data Structure (BIDS). So können Datensätze mit einer für alle verständlichen Struktur veröffentlicht und geteilt werden. Gorgolewski et al. (2016)
Hier sehen Sie ein Beispiel, wie ein fMRI Datensatz in BIDS Struktur umgewandelt wird:
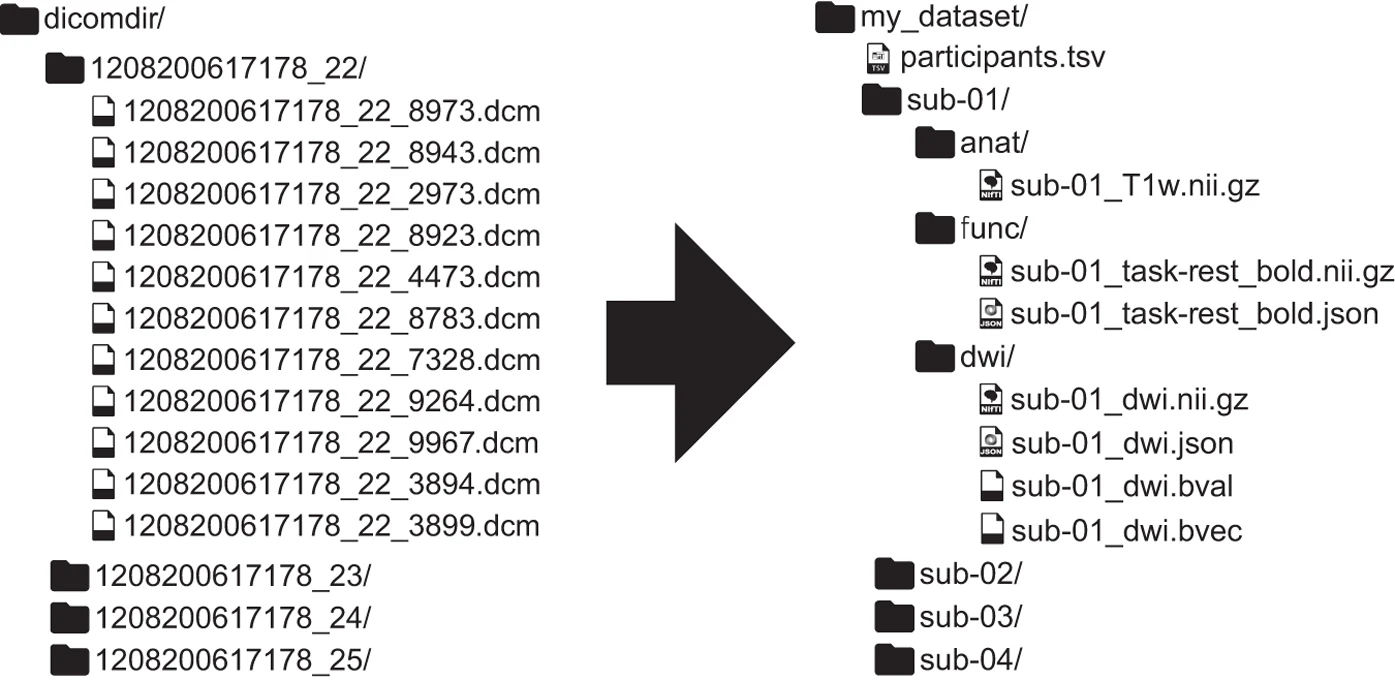 Gorgolewski et al. (2016)
Gorgolewski et al. (2016)
Coding: Best practices
Für das Veröffentlichen von Analyseskripts eignen sich Formate wie RMarkdown in R, oder LiveScripts in MATLAB aber auch .r-Skripte sehr gut. Beim Teilen von Code erhöht sich die Reproduzierbarkeit, wenn dieser verständlich strukturiert und kommentiert ist.
Wichtig ist hier:
Das Verwenden relativer Pfade (
data/raw_data.csvstattC:/Users/IhrName/Desktop/Projekt/Versuch1/finaleDaten/raw_data.csv)Konsequentes Löschen “alter” Variablen sowie Testen, ob der Code in sich läuft
Versionskontrolle: Entweder konsistentes Umbenennen oder mittels Github/Gitlab o.ä., Erstellen von Changelogs
Vor dem Veröffentlichen, lohnt es sich jemanden den Code ausführen lassen. So zeigt sich wo noch unklare Stellen sind, die Kommentare benötigen.
Beim Kommentieren von Code sollte folgendes beachtet werden:
Kommentare sollten geschrieben werden, wenn der Code erstellt wird und laufend überarbeitet werden. Oft wird es sonst nicht nachgeholt.
Wenn man nicht genau kommentieren kann, was man im Code macht, dann ist evtl. der Code unklar, oder man versteht ihn noch nicht. Vielleicht kann man Variablennamen vereinfachen/präzisieren und es braucht weniger Kommentare?
Wenn Code von anderen kopiert wird, sollte die Quelle angegeben werden.
Datenmanagement
In diesem Kapitel wird auf die Sicherstellung der Datenqualität, sowie die Wichtigkeit von Data collection plans und Data dictionaries eingegangen.
Datenqualität
Die folgenden sieben Indikatoren für gute Datenqualität wurden von C. Lewis (2024) zusammengefasst und sind hier zu finden.
1. Analysierbarkeit
Daten sollten in einem Format gespeichert werden in welchem sie analysiert werden sollen.
Die Variablennamen stehen (nur!) in der ersten Zeile.
Die restlichen Daten sind Werte in Zellen. Diese Werte sind analysierbar und Informationen sind explizit enthalten (keine leeren Felder, keine Farben als Information, nur 1 Information pro Variable).
2. Interpretierbarkeit
Variablennamen maschinenlesbar (kommen nur einmal vor, keine Lücken/spezielle Sonderzeichen/Farben, beginnen nicht mit einer Nummer, nicht zu lange)
Variablennamen sind menschenlesbar (klare Bedeutung, konsistent formattiert, logisch geordnet)
Datensätze sind in einem nicht-proprietären Format abgespeichert (z.B.
.csvnicht.xlsxoder.sav)
3. Vollständigkeit
Keine Missings, keine Duplikationen
Enthält alle erhobenen Variablen
Erklärung für alle Variablen in zusätzlichem File (z.B. in einem data dictionary)
4. Validität
Variablen entsprechen den geplanten Inhalten, d.h. Variablentypen und -werte stimmen mit Daten überein (z.B. numerisch/kategorial, range: 1-5)
Missings sind unverwechselbar angegeben (z.B. nicht -99, sondern NA)
5. Genauigkeit
Daten sollten in sich übereinstimmen
Daten sollten doppelt kontrolliert werden, wenn sie eingegeben werden
6. Konsistenz
Variablennamen sollten konsistent gewählt werden
Innerhalb wie auch zwischen den Variablen sollten Werte konsistent kodiert und formattiert sein
7. Anonymisiert
Alle Identifikationsmerkmale, sowohl direkte (Name, Adresse, E.Mail oder IP-Adresse, etc.) sowie indirekte (Alter, Geschlecht, Geburtsdatum, etc.) sollten so anonymisiert/kodiert sein, dass keine Rückschlüsse auf Personen möglich sind
Besondere Vorsicht bei: Offenen Fragen mit wörtlichen Antworten, extremen Werten, kleinen Stichproben/kleinen Zellgrössen (<3), Diagnosen, sensiblen Studienthemen
Es empfiehlt sich vor der definitiven Abspeicherung oder gar Veröffentlichung des Datensatzes, diese Punkte durchzuarbeiten und zu kontrollieren.
Data collection plan
Die Datenqualität kann erhöht werden, indem die obenstehenden Punkte schon bei der Planung der Datenerhebung beachtet und einbezogen werden. Wenn die Daten schon so eingegeben werden, werden die darauffolgenden Data Cleaning Schritte verkürzt und weniger Fehler passieren. Die Dateneingabe und -analyse sollte vor der Datenerhebung getestet und währenddessen immer wieder kontrolliert werden.
Data dictionaries
Ein data dictionary ist eine Sammlung von Metadaten die beschreibt, welche Variablen ein Datensatz enthält und wie die Werte dieser Variable formattiert sind.
Wichtige Inhalte für die Anwendung in den Neurowissenschaftlichen Forschung sind z.B. Name, Definition, Typ, Wertebereich (values), angewandte Transformationen, Geltungsbereiche (universe) und Format fehlender Angaben (skip logic).
Beispiel Data Dictionary
| variable | label | type | values | source | transformations | universe | skip_logic |
|---|---|---|---|---|---|---|---|
| participant identification | id | character, factor | “sub-001” - “sub-100” | raw files | anonymized variable “participant” | all | NA |
| participant age | age | integer | 18 - 55 | raw files | all | NA | |
| experimental block | block | integer | 1, 2 | raw files | all | NA |
Erstellen Sie in kleinen Gruppen für eines unserer beiden Experimente ein Data Dictionary. Verwenden Sie hierfür die in den oben abgebildeten Beispielstabelle vorhandenen Spalten.
Tipp: Sie können die Tabelle auch ganz simpel in RMarkdown erstellen in dem Sie die Tabelle als Text (nicht im Codechunk) erstellen. Wie das funktioniert können Sie hier nachlesen.
Informationen zu Datentypen finden Sie hier oder in der untenstehenden Tabelle
Datentypen
| Datentyp | Beschrieb | Beispiel |
|---|---|---|
| integer | Ganzzahliger Wert | 1, 2, 3, 29 |
| double, float | Zahl mit Kommastellen | 1.234, 89.3, 1.0 |
| character, factor | Text oder Kategorie (nicht numerisch interpretiert) | speed, 0 |
| string | Text oder Kategorie in “” oder ’’ | “sub-001” |
| datetime | Datum, Zeitpunkt | 2024-04-13, 21.12.23, 00:45:45 |
| boolean | Einer von 2 möglichen Werten | FALSE, 0 |
References
Reuse
Citation
@online{wyssen,
author = {Wyssen, Gerda and Fitze, Daniel},
title = {Good {Practices} in Der {Datenverarbeitung}},
url = {https://kogpsy.github.io/neuroscicomplabFS24//pages/chapters/goodpractices_data.html},
langid = {en}
}
